Python——提取Raw格式视频指定像素时序数据并做成数据集
前言
最近在尝试用时序方法对实验室的一些数据进行分类。由于没有通用的公共数据集,所以需要将实验数据做成一个小数据集,但是我找不到类似的开源代码来实现这个过程。自己写一个就行了。这部分代码主要实现:
- 读取raw格式的多张图像(如:240×360×1814就是大小为240*360共1814帧的数据)
- 通过不同的掩码提取不同类型的数据
- 处理为Pytorch dataloader函数可读取的形式
- 若无需进行深度学习任务也可提取其中的子类(类似于MATLAB中的矩阵的形式)方便其他任务操作
提示:后续有示例可供参考
1.过程可视化
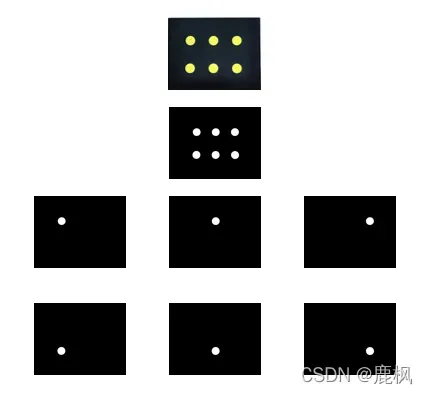
1.制作掩膜

左图为可见光实拍图;左边是左边黑色区域图像分割的结果;中间是面具;右二为实验数据中的一帧(全程位置不变);右边是模板按照右边第二个做图像配准,白色区域是某种类型。
注意:如果孔不是对称结构那得在最前面加一步镜像 毕竟实验数据是相当于反着拍的
2.用掩膜提取数据

3.其他类型数据重复上述流程
4.提取数据随便看看
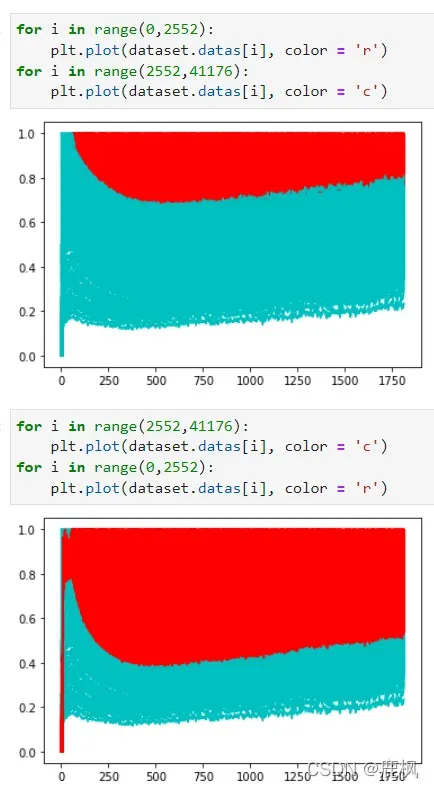
示例:pandas 是基于NumPy 的一种工具,该工具是为了解决数据分析任务而创建的。
2.代码
代码如下(示例):
import torch
import torch.utils.data
import os
import numpy as np
import cv2
class MyDataset(torch.utils.data.Dataset):
"""
读取raw图,乘上掩膜
把掩膜对应的部分做成数据集
不同的实验或者材料需要多调用几次这个类放在不同的data和dataloader里 最后再多调用几次triphard?
loss那里加权一下?
所以需要给[raw图地址, mask地址, label]作为一组输入 mask记得不同实验数据要做不同的配准
后续进行train和test的分割 trip组输入需要吗?
是否进行归一化 哪种归一化
Returns:
[sample, label]
"""
def __init__(self,
*arg, #这里把defect缺陷部分放进去 不一定有几种
train, #if true train set, or test set
Normalization,#是否需要对数据进行归一化,true就归 否则不归 具体归一化方法自己想想 可以改
normal
):
self.train = train
self.Normalization = Normalization
self.normal = normal
self.defect = arg
self.samples, self.labels = self.__getsamples()
def __readRawAndMask(self, data):#读取raw图和其对应的mask图 返回他们的乘积和label
rawPath, maskPath, label = data
# 利用numpy的fromfile函数读取raw文件,并指定数据格式
img_raw=np.fromfile(rawPath, dtype='uint16')
# 利用numpy中array的reshape函数将读取到的数据进行重新排列。 python的和MATLAB的排序方式不一样
img_raw=img_raw.reshape(320, 240, -1, order="F")##!!!!!!!!!!这里根据不同机器的输出大小进行修改
img_raw = img_raw.swapaxes(0,1)
#判断视频长度
self.length = img_raw.shape[2]
mask = cv2.imread(maskPath, 0)
#ret,mask = cv2.threshold(mask,127,1,cv2.THRESH_BINARY)#原本想二值化并reshape后和原图相乘 但出来的是三维 要二维
#mask = mask.reshape(240, 320, 1)
mask = np.array(mask , dtype = bool) #将mask由np型数组转换为布尔型数组方便后面掩膜
#mask = np.tile(mask, (1, 1, length))#原本想把mask拓展成三维的,发现输出变成一维向量了
data_roi = img_raw[mask,:]#把mask对应的部分输出
label = np.ones(data_roi.shape[0]) * label
label = np.expand_dims(label, 0)#这里拓展是为了方便后面做纵向拼接 记得 squeeze掉
print(label.shape)
return data_roi, label
def __getsamples(self):#用来把上面的乘积和label变成数据集
#取出数据
normalData, normalLabel = self.__readRawAndMask(self.normal)
for i in range(len(self.defect)):
locals()['defectData'+str(i)],locals()['defectLabel'+str(i)] = self.__readRawAndMask(self.defect[i])
#合并各类
defectData = locals()['defectData'+str(0)]
defectLabel = locals()['defectLabel'+str(0)]
if len(self.defect) > 1:
for i in range(len(self.defect)-1):
defectData = np.concatenate((defectData,locals()['defectData'+str(i+1)]),axis=0)#按行合并
defectLabel = np.concatenate((defectLabel,locals()['defectLabel'+str(i+1)]),axis=1)#按列合并
datas = np.concatenate((defectData, normalData),axis=0)#按行合并 每一行代表着一个样本
labels = np.concatenate((defectLabel, normalLabel),axis=1)#按列合并 每一列代表着一个样本对应的标签
labels = np.squeeze(labels)
print('总样本数量为:{}'.format(labels.shape))
#根据归一化的参数判断是否需要归一化 如有其他处理需要 可添加
if(self.Normalization):
datas = (datas - datas.min(axis=1, keepdims = True)) / (datas.max(axis=0) - datas.min(axis=1, keepdims = True))
#需要使样本数量均衡 triplethard这种形式需要均衡吗?
( 如有需要这里添加相应代码 )
#数据集并分割为训练集和测试集
#要不直接用另一个件当做测试?
( 如有需要这里添加相应代码 )
return datas, labels
def __len__(self):
num = self.labels.shape[1]
return num
def __getitem__(self, idx):
#这里不确定dataloader能不能用 没测
return torch.from_numpy(self.samples[idx]), torch.from_numpy(np.expand_dims(self.labels[idx], 0))
3.使用
代码如下(示例):
defectAll = [r'../data/花纹碳纤维板(8和6孔)/脉冲实验(好像)/Ht6.raw', '../data/缺陷mask_all.png', 1]
defect_0_0 = [r'../data/花纹碳纤维板(8和6孔)/脉冲实验(好像)/Ht6.raw', '../data/缺陷mask_0_0.png', 2]
normal = [r'../data/花纹碳纤维板(8和6孔)/脉冲实验(好像)/Ht6.raw', '../data/非缺陷mask.png', 0]
dataset = MyDataset(
defectAll,
defect_0_0,
train=True,
Normalization=True,
normal=normal#突然发现这里干了件蠢事 normal为什么要用成keyword argument,算了 清晰点也好
)
输出:
(1, 38624)
(1, 2552)
(1, 432)
总样本数量为:(41608,)
其他一些用途:
这里如果不用做深度学习训练的话只想要数组直接按照“一:4.图片中的代码提取子类即可 为np.array格式”,
最后:
如果不需要输出成tensor格式可以把
return torch.from_numpy(self.samples[idx]), torch.from_numpy(np.expand_dims(self.labels[idx], 0))
改成
return self.samples[idx], self.labels[idx]
顺便说一句,把顶部
import torch
import torch.utils.data
把它删掉,估计可以不用任何环境运行。
哦 OpenCV得装 或者 换别的自带包也行 不懂就在环境里
conda install opencv?大概?
具体顺序可以百度
文章出处登录后可见!
已经登录?立即刷新
