题目&作者
题目:一种用来做亚断层图像平均的特征引导的、聚焦的三维信号置换方法
作者:四位来自Stanford University,还有来自北大的郭强研究员
通讯:Axel T. Brunger (德裔美国生物物理学家、分子和细胞生理学家,斯坦福大学的神经学和神经科学教授)
摘要
(背景)
电子显微镜仪器、冷冻电子断层扫描数据收集和亚断层图像平均技术的进步使得分子及其复合物在其原生环境中的原位可视化成为可能。
(问题)
当前的数据处理流程通常将 subtomograms 提取为立方子体积,其关键假设是所选的感兴趣对象与其周围环境是离散的。然而,在对象处于其原生环境中的情况下,周围的密度可能会对随后的对齐和细化过程产生负面影响,导致由于未对齐而导致信息丢失。例如,来自周围膜的高密度可能主导膜蛋白的排列过程。
(本文工作)
在这里,我们开发了用于特征引导的 subtomogram 对齐和用于 subtomogram 平均的 3D 信号置换的方法。我们的 3D 信号置换方法随机化和过滤任何形状蒙版之外的体素,并模糊封装感兴趣对象的蒙版边界。随机化保留了全局统计属性,例如体素密度值的平均密度和标准偏差,有效地产生了围绕感兴趣对象的无特征背景。这种信号置换过程可以重复应用,其中包括 3D 信号置换子体积的中间对齐、掩模的居中以及掩模形状的可选调整。
在一个新的处理流程中实现了这些方法,流程从 tomograms 开始,包含特征引导的子断层图像提取和对齐、3D 信号置换和子断层图像可视化工具。例如,特征引导的对齐和 3D 信号置换可以改进其原生环境中突触蛋白复合物数据集的亚断层平均图。
Introduction
(背景介绍,引出问题)
低温电子显微镜 (cryoEM) 仪器、数据收集和处理方面的进步使我们能够在有利的情况下以原子分辨率确定单粒子结构。 特别是,直接电子探测器的发展允许通过提高探测量子效率(DQE)收集更高分辨率的信息,并且校正电子束引起的漂移提高了分辨率。
单粒子 cryoEM 主要使用纯化的大分子进行,省略了细胞环境,为了在其原生环境中研究这些大分子,通常使用由一系列倾斜系列组成的低温电子断层扫描 (cryoET)。 原则上,从 cryoET 获得的 3D 重建允许分割和随后对齐特征密度,如在他们的原生环境中的膜、细丝或大分子复合体。随后,可以提高感兴趣对象的分辨率,并通过亚断层图平均减轻丢失楔的影响,在有利的情况下达到亚纳米分辨率。子断层图像平均通常需要收集许多断层图像。收集此类大型 cryoET 数据集通常具有挑战性,部分原因在于必要的数据收集时间(每个断层图像最多一小时)。自动 cryoET 数据收集和光束图像偏移的改进减少了数据收集时间,仪器的进一步进步最终将允许在未来连续进行 cryoET 数据收集。图像处理和亚断层图像平均方法的改进也导致了大分子复合物在其原生环境中的更高质量图像。
然而,即使有了这些进步,对于感兴趣的对象处于拥挤环境中的情况,亚断层平均仍然具有挑战性。在这些情况下,许多对象可能会被自动化程序错位,导致在最佳情况下分辨率较低,而在最坏情况下完全丢失或平均形态特征。增加断层图像的数量,从而增加对象数量,是解决这个问题的一种方法,但这种策略的效果从根本上受到样品制备和数据收集时间的限制。这种错位问题对于跨膜蛋白来说尤其令人烦恼,其中由膜贡献的高强度体素会影响图像对齐和分类。
因此,需要开发计算方法,以最大化有关感兴趣对象的所需信息,这些信息可以通过从 cryoET 数据集中通过亚断层图平均获得。
(介绍现有 STA 算法)
当前可用的用于亚断层图平均的软件包和管道(Bharat 等人,2015 年;Bharat 和 Scheres,2016 年;Chen 等人,2019 年;Galaz-Montoya 等人,2016 年;Tegunov 等人,2021 年;Tegunov 和 Cramer,2019 年; Wan et al., 2020) 包括 CTF 确定、CTF 校正和亚断层图提取、平均、分类和 3D 细化。通常,分类和 3D 细化功能已从单粒子 EM 方法中采用,其中假设具有低噪声周围背景的离散目标对象。这种方法可能与非球状连续物体不兼容,例如与膜相关或嵌入膜中的蛋白质。在细化过程中对 3D 类平均密度图的简单掩蔽仅部分解决了不良背景信息的问题,因为这种掩蔽实际上并没有从负责偏置分类和对齐的 3D 断层扫描重建中去除体素。因此,随着细化的进行,这些高度影响的背景体素可以重新进入被掩盖的感兴趣区域并进行偏差细化。对于单粒子 cryoEM 数据集,可以通过在 2D 投影图像中通过部分减去此类密度来去除周围密度的贡献来执行信号减法。然而,在 cryoET 数据集的 2D 倾斜图像中修改附近的密度可能不是最佳的。因此,3D 断层扫描重建中的信号修改对于亚断层图像平均应该更成功。
(介绍本文贡献)
开发了一种新的 cryoET/subtomogram averaging 流程(CreSTA),包括特征引导对齐和通过断层重建的信号置换来修改 3D 信号的新方法。
3D 信号置换方法随机化了一个可以是任何形状并且模糊了掩码边界的掩码外的体素。此外,提供了过滤掩模外的随机体素的选项,以减弱可能由膜密度引起的明显异常值。该方法有效地在 3D 中产生围绕感兴趣对象的无特征背景。这种信号置换过程可以重复应用,其中包括 3D 信号置换子体积的 3D 细化、掩模的中心化和掩模形状的可选调整。
CrESTA 从 3D 断层扫描重建开始(包括但不限于使用 NovaCTF 进行 CTF 校正的断层扫描重建),并包含特征引导的子断层图像对齐、提取、信号置换和子图可视化工具。
对在原生环境中的突触蛋白复合物的应用说明了 CrESTA 如何改进对亚断层图平均数据的处理。
Methods
探讨了 subtomogram 的对齐策略,阐述了 3D 信号置换的实用性。
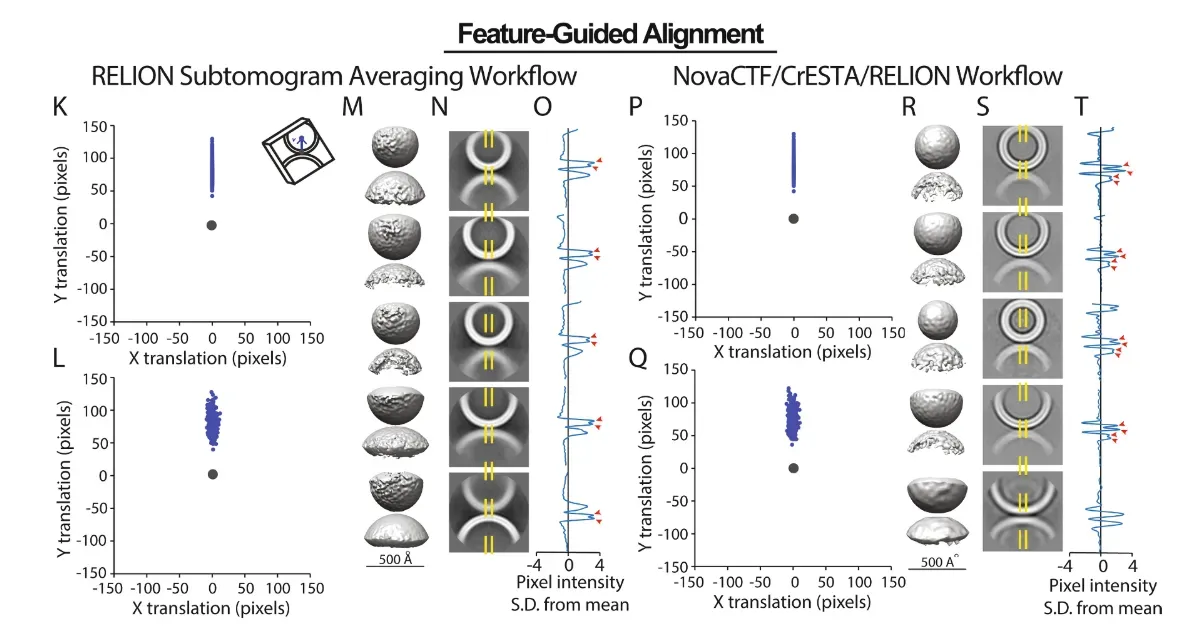
- 对比了两种用于初始对齐的方法:
free alignment和feature-guided alignment,如图1。 - 此外,评估了在
NovaCTF/ CrESTA/RELION工作流中实现的3D信号置换方法(图2和图3)对亚层析平均的影响。
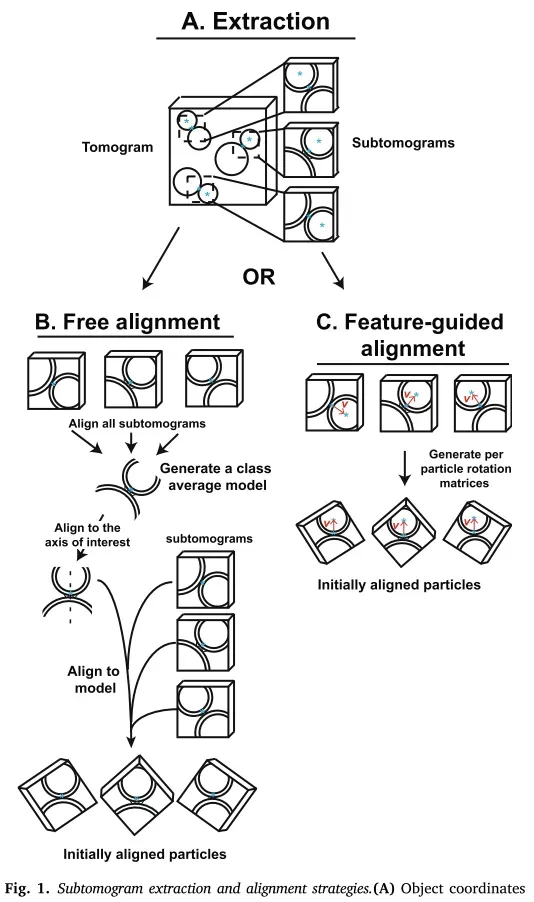
B. 在自由对齐中,首先对齐所有子断层图以生成类平均模型。 然后将模型手动旋转到感兴趣的轴(虚线)。 然后将子断层图与这个手动旋转的类平均模型对齐,以产生最初对齐的粒子。
C. 特征引导对齐利用第二个对象坐标的选择,这里是 ISV 中心坐标(囊泡中心的蓝色星号)。 然后使用 ISV 膜和中心坐标之间的向量 v 来对齐所有 subtomograms(红色箭头,这里将所有囊泡-囊泡对与顶部的 ISV 囊泡对齐)
示例数据集
对突触蛋白数据集进行了介绍。(synaptic protein dataset)
电子断层重建
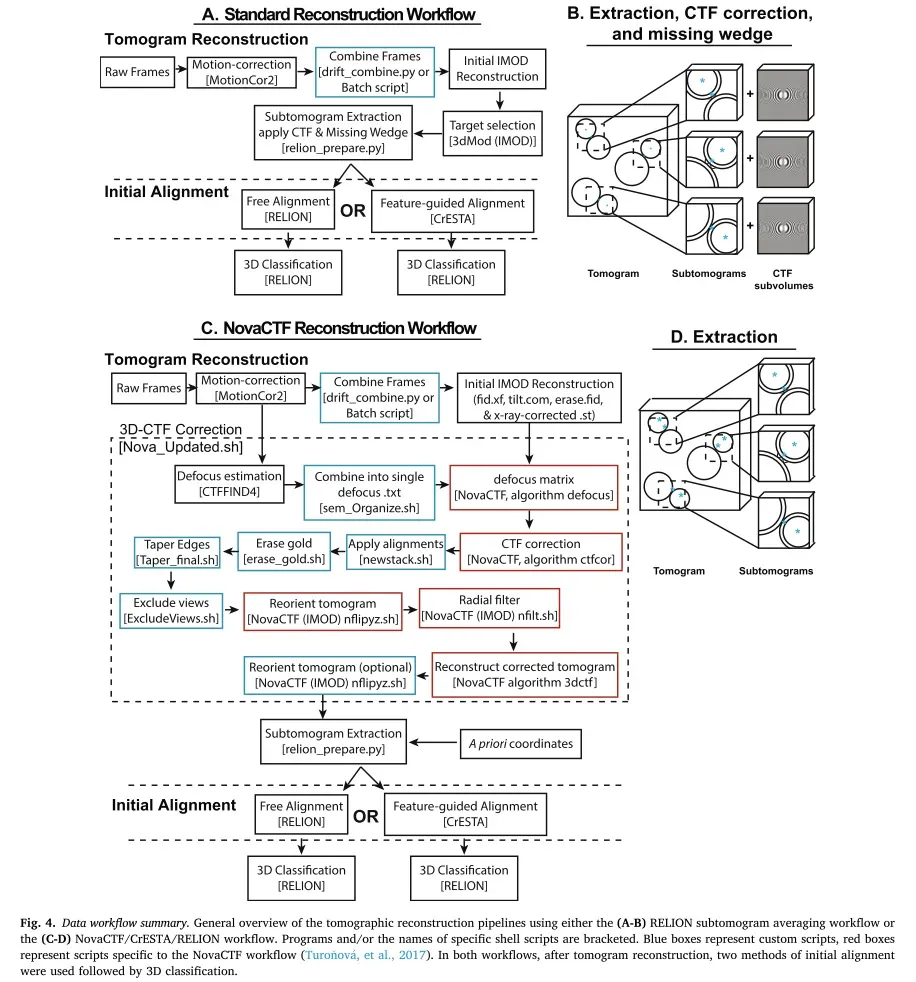
本文使用了两种断层重建和三维CTF矫正方法,来测试两种对齐策略:
- RELION-3;(图中 A、B)
- (Ours) NovaCTF/ CrESTA/ RELION:流程和 STOPGAP 相似,如图4。
对于 3D 信号置换,必须计算 3D CTF 校正的断层重建。 此外,全 3D CTF 校正对于倾斜系列来说是必不可少的,因为在倾斜系列的外围有 >1 µm 的散焦差异,或在散焦估计中高达 100% 的误差。 我们使用了 NovaCTF 对整个断层图进行 3D CTF 估计和校正。 NovaCTF 可以通过相位翻转或 CTF 乘法来执行 CTF 校正。 对于我们的突触蛋白 cryoET 数据集,我们使用了相位翻转。
提取 subtomograms
对于突触蛋白数据集,本文执行了两个单独的子断层图提取。
- 第一次提取产生突触界面子断层图:即整个囊泡-囊泡“突触”的子断层图。使用手动选择的 ISV 膜坐标(即,最接近相互作用的 SM 囊泡的 ISV 膜的点;图 1A,膜中的星号)提取突触界面子断层图,用于 RELION 子断层图平均和 NovaCTF/CrESTA/RELION重建工作流程。 ISV 可以很容易地通过存在属于囊泡 ATP 酶的大蛋白质密度来识别(图 3A、3B)。为了跟踪和可视化囊泡界面的对齐,我们选择了 ISV 中心的第二个点(图 1A,囊泡中心的星号)。
- 第二次提取侧重于突触界面处存在的单个膜间密度(即突触蛋白复合物密度)。对于第二次提取,通过对 Chimera 中的 Wiener 过滤突触界面亚断层图的视觉检查获得坐标(图 3D,右)。手动选择各个膜间密度的近似中心坐标。

CTF 矫正和缺失楔
RELION subtomogram 平均工作流程包括一个 3D CTF 校正,它使用 CTFFIND4 估计散焦,并在每个子断层图像的基础上校正 CTF。 3D CTF 校正针对每个子断层图像执行——更准确地说,它对特定子断层图像中的所有体素应用相同的 CTF 校正参数,尽管体积不同部分的散焦发生变化。 CTF 校正应用于傅立叶空间,它还包含丢失楔的影响,并包括剂量依赖的 B 因子模型。 对于 CTFFIND4 可能无法为某些数据集(包括突触蛋白数据集)产生可靠估计的高倾斜图像,散焦值设置为平均散焦估计。对于 NovaCTF 生成的 CTF 校正断层扫描重建,在随后的 RELION 分类和细化过程中仍需要考虑缺失楔的影响。 对于突触蛋白数据集,使用 tom_wedge.m (图 3C) 生成了一个合成的单个缺失楔体积,并且该体积被指定为每个子断层图像的“_rlnCtfImage” ,在输入到 RELION 的 STAR 文件中。 原则上,这种方法也可以推广到应用 dose filtering。
subtomograms 的初始对齐
由于每个目标密度的可能方向有很大的样本空间,即便用适度数量的子断层图来执行完全无偏、无参考的 3D 类平均计算要求也很高。 此外,在极性伪对称的情况下,这种无偏无参考的类平均可能不会成功。 限制可搜索样本空间的一种方法是首先对齐所有子断层图,以便感兴趣的对象以相似的方向开始。 这里,评估了初始对齐的两种方法:自由对齐和特征引导对齐。
自由对齐
使用了两步的方法来进行亚层析成像的初始无参考对齐。第一步使用RELION的三维细化功能在没有参考的情况下对亚层析图像进行对齐。使用一个类对亚层析图进行对齐和平均,无参考,对平移和角度采样也没有限制。
通常生成的平均 3D map 处于任意方向。为了更好地控制后续的重新对齐(并将结果与特征引导对齐进行比较),我们将初始对齐转换为标准方向。用CrESTA的可视化工具显示初始自由对齐的平均值。然后手动旋转,使感兴趣的对象可以任意对齐到一个轴。在第二步中,用这个旋转过的average map 作为初始参考,对一个类进行另一个3D RELION细化。这个重新对齐步骤的结果是,所有子层析图都是定向的,以便围绕这个感兴趣的轴旋转对齐。
特征引导的对齐
通常通过选择感兴趣对象中心的单个坐标,然后在其周围提取立方子体积来获得子断层图。这种坐标选择可以通过使用IMOD、EMAN2等其他包的手动检查来完成,或者通过模板匹配获得。这些中心坐标没有方向信息。在可以使用有关对象方向的其他上下文的情况下,利用此信息可以降低后续细化步骤中的自由度。此外,在具有极性的伪对称(例如,夹在两个不同的脂质双层之间的结构)的情况下,子断层图可能会变得不正确。
为了减少这种错误,可以采用特征引导的对齐方法。我们开发了一种特征引导对齐方法,其中为每个感兴趣的对象(除了中心坐标)手动选择第二个坐标,例如与膜蛋白相关的特定膜。当然,这要求可以通过目测明确地确定膜的身份。该方法应该适用于具有上下文信息的细胞子结构,例如单膜上的膜蛋白或与细丝相关的蛋白质。然后使用两个选定坐标之间的向量将感兴趣的轴与任意选定的 z 轴对齐。
在特征引导对齐之后,建议执行 3D 分类/ restrained refinement,有效地在向量 v (两个坐标之间形成的向量)周围施加受限旋转搜索。
3D 信号置换和 subtomograms 的过滤
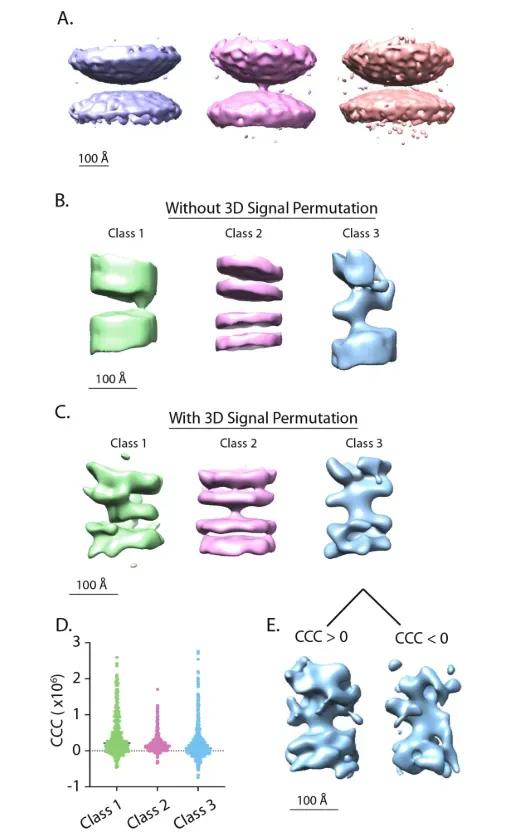
我们在NovaCTF/CrESTA/RELION工作流中开发了一种3D信号置换方法,以消除子断层图中感兴趣对象周围潜在干扰的背景体素(图3)。
首先,使用“创建遮罩”模块在 CrESTA 内生成简单的几何二元遮罩M(例如,球体或圆柱体,取决于感兴趣对象的形状),遮罩内外的值分别设置为1和0。在掩码M用于3D信号置换之前,将来自每个子图的初始对齐的旋转矩阵R和平移向量T反转,并在每个子图的基础上应用于该掩码。
然后,脚本通过使 mask 内的体素索引不受影响并随机排列
mask 外的体素索引,在子图体积
上执行3D信号置换,从而生成3D信号置换体积
。
for voxels
inside
for voxels
outside
M掩模外体素指数的排列移除了掩模区域外的特征,而不改变整个子断层图体素的全局统计信息(平均值和标准偏差)。
为了减少遮罩边缘边界的影响,仅在遮罩外部排列随机选择的体素子集,作为距遮罩边界距离的函数。随着距离遮罩边界的距离增加,随机排列的体素数量增加。 因此,信息减少取决于到遮罩边界的距离。计算具有定义距离范围(伪壳)的一组体素的问题通过将给定遮罩“增长”到指定数量来解决。将重复生长遮罩和体素置换的过程,直到最终置换生长遮罩外的所有体素。
如果背景包含偏移分类的体素(即,体素强度分布两端的极值),则过滤这些体素是有用的。
我们提供了一个可选的过滤器,它用从正态分布范数 中随机选择的值替换强度非常大或非常小的体素(由用户定义的平均值m的标准偏差w,b来确定),这些值的平均值为m,标准偏差为s(m通常设置为0,s设置为1)。此高强度过滤器保持体素平均值,仅细微更改体素标准偏差,同时进一步降低异常背景体素的影响。
图3E是在膜周围的3D信号置换的子断层图示例。信号置换和滤波的子图可以输入到 RELION 或其他程序中,以便使用初始对齐的方向信息进行3D分类/细化。
重复 3D 信号置换
在每一类平均值收敛后,可以执行新一轮的3D信号置换。该信号置换再次在原始子图上执行,但使用先前3D细化的对准信息,而不是手动选择的坐标和初始对准。此步骤有助于纠正初始坐标拾取中的不准确。
此外,如果在该阶段类平均值基本上是异质的,则为每个类使用用于3D信号置换的唯一几何掩码可能是有益的,并且可以同时进行。例如,从先前的对齐/优化作业中获得的类平均映射可用于生成更明确的掩码。然而,评估此类遮罩可能引入的潜在偏差非常重要,例如通过交叉验证,例如,出现不属于遮罩一部分的新特征。使用这些校正和改进的3D信号置换子图执行后续对齐/细化。根据这些类别平均值的质量,可以根据需要多次重复由3D信号置换和3D细化完成的子断层图细化和唯一掩模生成过程。
用 CrESTA 手动和基于互相关修剪类、可视化 subtomograms
Results
自由对齐
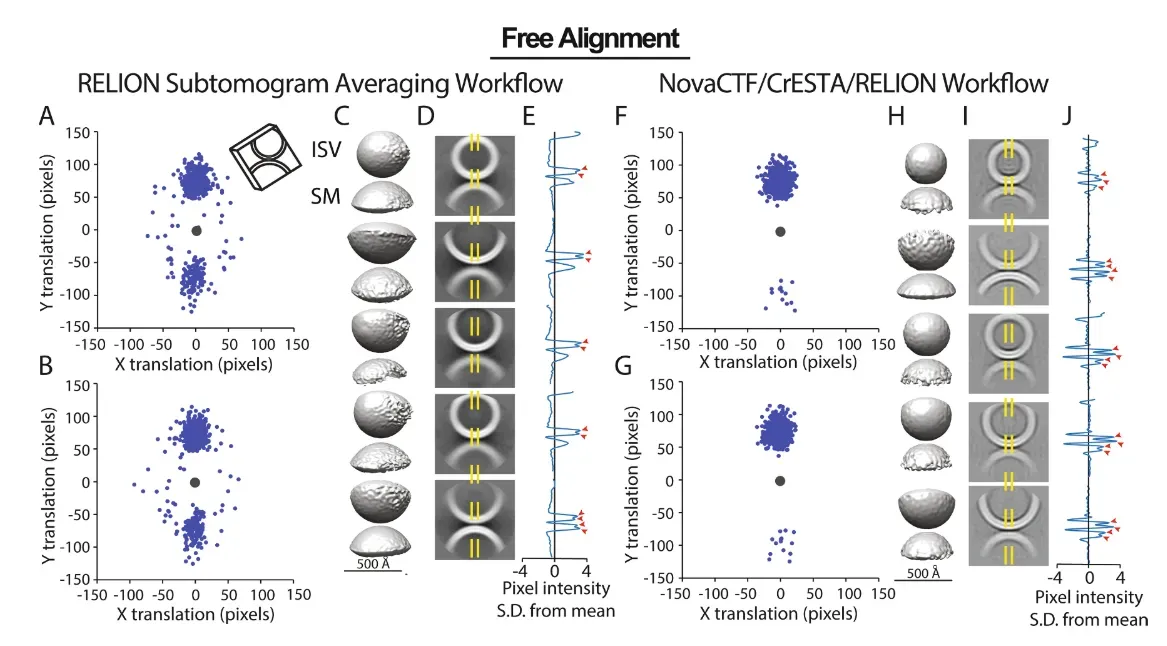
由于所有ISV中心坐标预计都属于顶部群集,因此自由对齐过程产生了大量错误对齐的子集。除了翻转之外,一些ISV中心还存在一些水平扩展,表明接口倾斜不当。
当使用NovaCTF/ CrESTA/RELION工作流时,可以实现更好的自由对齐。与RELION次层析图平均工作流相比,初始对齐后翻转明显减少。
特征引导的对齐

3D 信号置换和密度过滤
Conclusions and Discussion
子断层图平均的一个关键限制可能是感兴趣的对象数量少,这一问题由于收集断层图像的时间密集型过程以及对象的潜在拒绝或错位而更加复杂。因此,优化数据处理、子图对齐和平均工作流程对于提高最终分辨率至关重要。理想情况下,每个感兴趣的对象最终都应该正确地对齐和分类,以最大限度地增加生成的类平均图的信息内容。我们开发了一个新的子断层图平均工作流(NovaCTF/CrESTA/RELION),包括:
1)围绕公共轴的特征引导的子断层图提取和对齐,
2)使用几何和数据启发的掩模从子断层图中进行三维信号置换,并随机化周围的体素值,然后使用3D分类/v约束细化进行子断层图平均,
3)使用手动检查和互相关方法选择子集,
4)使用重新对齐/调整的掩码重复3D信号置换和v-约束细化。
除了像这里介绍的突触蛋白复合物这样的无细胞系统之外,CrESTA和NovaCTF/CrESTA/RELION工作流应该在其他生物系统中有用。例如,CrESTA可能在细胞层析成像中有用,其中感兴趣的对象更可能位于特征丰富的区域。此外,细胞环境为感兴趣的对象提供方向信息,特征引导对齐将利用此信息。例如,我们的方法应该普遍适用于所有膜-膜或细胞-细胞连接。
文章出处登录后可见!
