从今天开始正式进入针对药物发现的相关数据集的介绍与应用。文章中所有的代码都是依托PYG来实现的。
PPI数据集下载地址:https://data.dgl.ai/dataset/ppi.zip
1. 数据集介绍
蛋白质 – 蛋白质相互作用网络,包含位置基因集,基序基因集和免疫特征作为特征(共50个)和基因本体集作为标签(共121个)。
| graphs | nodes | edges | features | tasks |
|---|---|---|---|---|
| 20 | ~2,245 | ~61,318 | 50 | 121 |
# 加载数据集
dataset = PPI(root='E:/data/ppi')# 若是先前已经存在数据集,那么就把对应的数据集的文件放在以raw命名的文件夹中即可
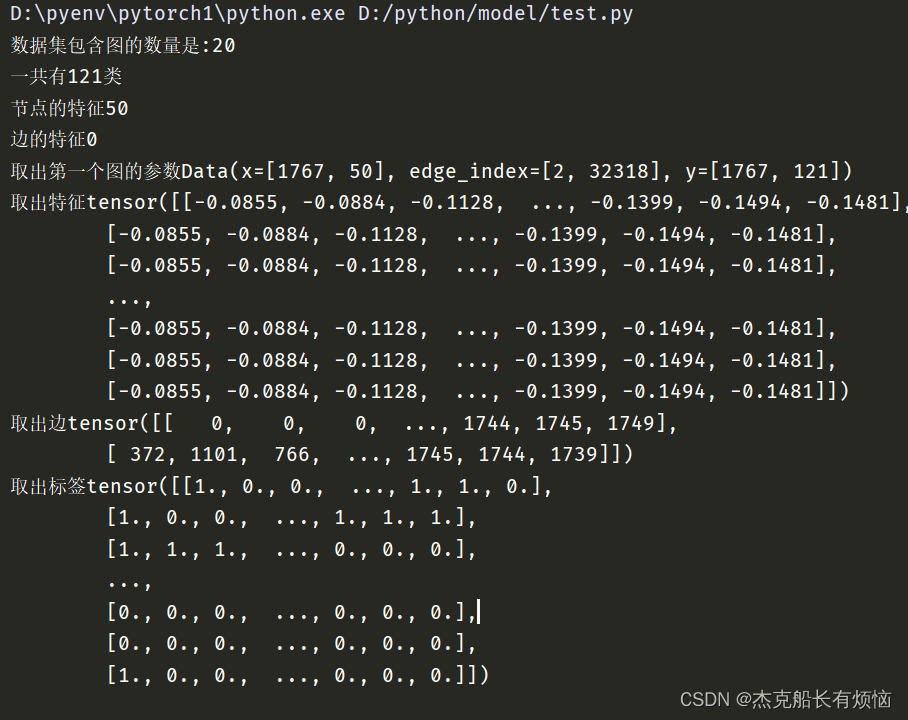
print(f'数据集包含图的数量是:{len(dataset)}')
print(f'一共有{dataset.num_classes}类')
print(f'节点的特征{dataset.num_node_features}')
print(f'边的特征{dataset.num_edge_features}')
print(f'取出第一个图的参数{dataset[0]}')
print(f'取出特征{dataset[0].x}')
print(f'取出边{dataset[0].edge_index}')
print(f'取出标签{dataset[0].y}')
2. 视图化PPI
2.1 networkx
from torch_geometric.datasets import PPI
from torch_geometric.utils import to_networkx
import matplotlib.pyplot as plt
import networkx as nx
# 加载数据集
dataset = PPI(root='E:/data/ppi')# 若是先前已经存在数据集,那么就把对应的数据集的文件放在以raw命名的文件夹中即可
#先取PPI中的第一个图来进行可视化
g = dataset[0]
g = to_networkx(g)
nx.draw(g,with_labels=g.nodes)
plt.show()
2.2 t-SNE
t-SNE是一种集降维与可视化于一体的技术,它是基于SNE可视化的改进,解决了SNE在可视化后样本分布拥挤、边界不明显的特点,是目前最好的降维可视化手段。
关于t-SNE的历史和原理详见 从SNE到t-SNE再到LargeVis (bindog.github.io)
2.2.1 参数
| parameters | 描述 |
|---|---|
| n_components | 嵌入空间的维度 |
| perpexity | 混乱度,表示t-SNE优化过程中考虑邻近点的多少,默认为30,建议取值在5到50之间 |
| early_exaggeration | 表示嵌入空间簇间距的大小,默认为12,该值越大,可视化后的簇间距越大 |
| learning_rate | 学习率,表示梯度下降的快慢,默认为200,建议取值在10到1000之间 |
| n_iter | 迭代次数,默认为1000,自定义设置时应保证大于250 |
| min_grad_norm | 如果梯度小于该值,则停止优化。默认为1e-7 |
| metric | 表示向量间距离度量的方式,默认是欧氏距离。如果是precomputed,则输入X是计算好的距离矩阵。也可以是自定义的距离度量函数。 |
| init | 初始化,默认为random。取值为random为随机初始化,取值为pca为利用PCA进行初始化(常用),取值为numpy数组时必须shape=(n_samples, n_components) |
| verbose | 是否打印优化信息,取值0或1,默认为0=>不打印信息。打印的信息为:近邻点数量、耗时、 |
| random_state | 随机数种子,整数或RandomState对象 |
| method | 两种优化方法:barnets_hut和exact。第一种耗时 |
| angle | 当method=barnets_hut时,该参数有用,用于均衡效率与误差,默认值为0.5,该值越大,效率越高&误差越大,否则反之。当该值在0.2-0.8之间时,无变化。 |
返回对象的属性表:
| Atrtributes | 描述 |
|---|---|
| embedding_ | 嵌入后的向量 |
| kl_divergence_ | KL散度 |
| n_iter_ | 迭代的轮数 |
2.2.2 转换后维度
tsne = manifold.TSNE(n_components=3,init='pca',random_state=2022)
x_tsne = tsne.fit_transform(labels.x)
print(f'原始的数据嵌入维度{labels.x[-1]}\n转换后维度嵌入{x_tsne[-1]}')
tsne = manifold.TSNE(n_components=2,init='pca',random_state=2022)
x_tsne = tsne.fit_transform(labels.x)
# print(f'原始的数据嵌入维度{labels.x[-1]}\n转换后维度嵌入{x_tsne[-1]}')
fig = plt.figure(figsize=(8, 8))
plt.scatter(x_tsne[:, 0], x_tsne[:, 1],c=label_onehot1, cmap=plt.cm.Spectral)
plt.show()
附则
由于PPI的标签信息是一个121位的二进制编码,这里所以就无法使用,下面是我将这个1767X121编码转化为1767X1的过程
重点感谢我的同学:万亮和钰杰。
因为他们的出谋划策,我才能顺利的完成这一小节的工作。
labels = labels.y.numpy()
np.savetxt('E:\data\ppi/label_onehot.csv',labels,fmt='%d')
label_onehot = np.loadtxt('E:\data\ppi/label_onehot.csv',dtype='int')
dic1={}
v=[]
# 建立一个可以将标签中相同节点的值放在一起的字典
for i in range(len(label_onehot)):
for j in range(len(label_onehot)):
if all(label_onehot[i]==label_onehot[j]):
v.append(j)
else:
continue
dic1[i]=v
v= []
# 去出字典中的重复项
dicv =[i for i in range(1767)]
for i in dicv:
for j in dicv:
if i != j and all(label_onehot[i] == label_onehot[j]):
dic1.pop(j)
dicv.remove(j)
# #找到值大于1的键
pa = []
for i in dicv:
if len(dic1[i]) >1:
pa.append(i)
# 对照字典里面的值对标签进行修改
dicp = [i for i in range(1767)]
labelp =pd.read_csv('E:\data\ppi/label_onehot1.csv')
labelp['index']=dicp
for p in pa:
labelp['index'].replace(dic1[p],p,inplace=True)
labelp.to_csv('E:\data\ppi/label_onehot2.csv')
label_onehot1 = np.loadtxt('E:\data\ppi/label_onehot3.csv',dtype=int)
文章出处登录后可见!
已经登录?立即刷新
