今天介绍Jinmiao Chen教授组的又一新作,今年八月投的稿,虽然还在审稿中,但是代码和论文都放出来了,跑了一下结果也是非常的好,甚是崇拜,拜读后过了总结一下。(其实之前也研读过SEDR那篇论文,同样精彩。)
论文名字是DeepST: A versatile graph contrastive learning framework for spatially informed clustering, integration, and deconvolution of spatial transcriptomics,模型总体思想是AE+DGI的组合,即总体的模型是一个图自编码器,这与STAGATE很相似,只是前者编码器用的GCN,后者GAT。损失函数即重构的基因表达数据和原始数据的MSE,然后加入了DGI的损失函数,即局部特征与全局特征的相似性。
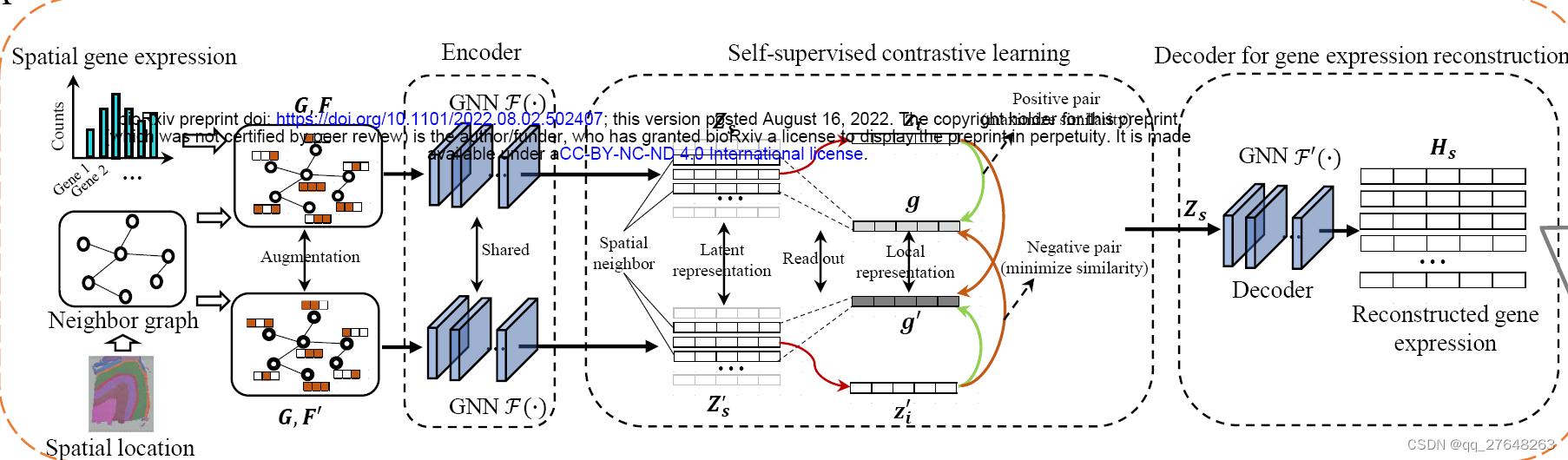
下面来看一下代码,还是以DLPFC 12个切片为例。前边的读取 聚类和STAGATE非常类似,包括数据读取、设置R的路径等
input_dir = os.path.join('Data', dataset)
adata = sc.read_visium(path=input_dir, count_file=dataset + '_filtered_feature_bc_matrix.h5')
adata.var_names_make_unique()
然后是数据的准备阶段。
model = DeepST.DeepST(adata, device=device)具体来看看里面的程序。首先是设置随机种子。python环境种子、随机种子、np库种子,cuda种子、卷积层预先优化等。
def fix_seed(seed):
os.environ['PYTHONHASHSEED'] = str(seed)
random.seed(seed)
np.random.seed(seed)
torch.manual_seed(seed)
torch.cuda.manual_seed(seed)
torch.cuda.manual_seed_all(seed)
cudnn.deterministic = True
cudnn.benchmark = False
os.environ['PYTHONHASHSEED'] = str(seed)
os.environ['CUBLAS_WORKSPACE_CONFIG'] = ':4096:8'
然后是对基因表达数据进行预处理。
def preprocess(adata):
sc.pp.highly_variable_genes(adata, flavor="seurat_v3", n_top_genes=3000)
sc.pp.normalize_total(adata, target_sum=1e4)
sc.pp.log1p(adata)
sc.pp.scale(adata, zero_center=False, max_value=10)多了一个scale,将数据缩放到单位方差和零均值。 不太明白sc.pp.scale和sc.pp.normalize_total是不是有重复呢,有懂的大神解答下。
然后是构建图的邻接矩阵。找到每个spot最近的三个spot认为是其有连接,边为1,其他的spot之间没有连接。
def construct_interaction(adata, n_neighbors=3):
"""Constructing spot-to-spot interactive graph"""
position = adata.obsm['spatial']
# calculate distance matrix
distance_matrix = calculate_distance(position.astype(np.float64))
n_spot = distance_matrix.shape[0]
adata.obsm['distance_matrix'] = distance_matrix
# find k-nearest neighbors
interaction = np.zeros([n_spot, n_spot])
for i in range(n_spot):
vec = distance_matrix[i, :]
distance = vec.argsort()
for t in range(1, n_neighbors + 1):
y = distance[t]
interaction[i, y] = 1
adata.obsm['graph_neigh'] = interaction
#transform adj to symmetrical adj
adj = interaction
adj = adj + adj.T
adj = np.where(adj>1, 1, adj)
adata.obsm['adj'] = adj然后构建正负类的标签。这里用到了DGI的思想,即通过随机变换特征的顺序构建负类,负类标签为0,正类为1。
def add_contrastive_label(adata):
# contrastive label
n_spot = adata.n_obs
one_matrix = np.ones([n_spot, 1])
zero_matrix = np.zeros([n_spot, 1])
label_CSL = np.concatenate([one_matrix, zero_matrix], axis=1)
adata.obsm['label_CSL'] = label_CSL随后构建模型和训练模型。
adata = model.train_DeepST()还是看看具体程序。首先是提取特征,基因表达数据就是把先前处理的数据拿出来即可,这里并没有任何操作。
def get_feature(adata, deconvolution=False):
if deconvolution:
adata_Vars = adata
else:
adata_Vars = adata[:, adata.var['highly_variable']]
if isinstance(adata_Vars.X, csc_matrix) or isinstance(adata_Vars.X, csr_matrix):
feat = adata_Vars.X.toarray()[:, ]
else:
feat = adata_Vars.X[:, ]
# data augmentation
feat_a = permutation(feat)
adata.obsm['feat'] = feat
adata.obsm['feat_a'] = feat_a feat_a是构建的负类,随机调换特征顺序,具体看下。
def permutation(feature):
# fix_seed(FLAGS.random_seed)
ids = np.arange(feature.shape[0])
ids = np.random.permutation(ids)
feature_permutated = feature[ids]
return feature_permutated然后就是构建模型和训练模型了,初始化参数和转化tensor就不放上了。下一步将adj邻接矩阵标准化构建GCN的邻接矩阵。
def normalize_adj(adj):
"""Symmetrically normalize adjacency matrix."""
adj = sp.coo_matrix(adj)
rowsum = np.array(adj.sum(1))
d_inv_sqrt = np.power(rowsum, -0.5).flatten()
d_inv_sqrt[np.isinf(d_inv_sqrt)] = 0.
d_mat_inv_sqrt = sp.diags(d_inv_sqrt)
adj = adj.dot(d_mat_inv_sqrt).transpose().dot(d_mat_inv_sqrt)
return adj.toarray()
def preprocess_adj(adj):
"""Preprocessing of adjacency matrix for simple GCN model and conversion to tuple representation."""
adj_normalized = normalize_adj(adj)+np.eye(adj.shape[0])
return adj_normalized 然后解卷积部分10x数据并没有,先不分析了,最后再单独看。程序到这数据处理部分就彻底结束了,在看模型构建。
self.model = Encoder(self.dim_input, self.dim_output, self.graph_neigh).to(self.device)然后看一下模型的具体构建,正样本经过两层GCN后生成编码数据emb,负样本经过相同的两层GCN后生成负样本编码数据emb_a。
class Encoder(Module):
def __init__(self, in_features, out_features, graph_neigh, dropout=0.0, act=F.relu):
super(Encoder, self).__init__()
self.in_features = in_features
self.out_features = out_features
self.graph_neigh = graph_neigh
self.dropout = dropout
self.act = act
self.weight1 = Parameter(torch.FloatTensor(self.in_features, self.out_features))
self.weight2 = Parameter(torch.FloatTensor(self.out_features, self.in_features))
self.reset_parameters()
self.disc = Discriminator(self.out_features)
self.sigm = nn.Sigmoid()
self.read = AvgReadout()
def reset_parameters(self):
torch.nn.init.xavier_uniform_(self.weight1)
torch.nn.init.xavier_uniform_(self.weight2)
def forward(self, feat, feat_a, adj):
z = F.dropout(feat, self.dropout, self.training)
z = torch.mm(z, self.weight1)
z = torch.mm(adj, z)
hiden_emb = z
h = torch.mm(z, self.weight2)
h = torch.mm(adj, h)
emb = self.act(z)
z_a = F.dropout(feat_a, self.dropout, self.training)
z_a = torch.mm(z_a, self.weight1)
z_a = torch.mm(adj, z_a)
emb_a = self.act(z_a)
g = self.read(emb, self.graph_neigh)
g = self.sigm(g)
g_a = self.read(emb_a, self.graph_neigh)
g_a = self.sigm(g_a)
ret = self.disc(g, emb, emb_a)
ret_a = self.disc(g_a, emb_a, emb)
return hiden_emb, h, ret, ret_a接下来是通过read函数构造全局特征,即将局部特征求均值就是全局特征,这里用了DGI的思想。
class AvgReadout(nn.Module):
def __init__(self):
super(AvgReadout, self).__init__()
def forward(self, emb, mask=None):
vsum = torch.mm(mask, emb)
row_sum = torch.sum(mask, 1)
row_sum = row_sum.expand((vsum.shape[1], row_sum.shape[0])).T
global_emb = vsum / row_sum
return F.normalize(global_emb, p=2, dim=1) 然后还是DGI的思想,希望正样本的局部信息与全局信息越相似越好,负样本的局部信息与全局信息越不相似越好门下面是求解相似性的分辨函数。
class Discriminator(nn.Module):
def __init__(self, n_h):
super(Discriminator, self).__init__()
self.f_k = nn.Bilinear(n_h, n_h, 1)
for m in self.modules():
self.weights_init(m)
def weights_init(self, m):
if isinstance(m, nn.Bilinear):
torch.nn.init.xavier_uniform_(m.weight.data)
if m.bias is not None:
m.bias.data.fill_(0.0)
def forward(self, c, h_pl, h_mi, s_bias1=None, s_bias2=None):
c_x = c.expand_as(h_pl)
sc_1 = self.f_k(h_pl, c_x)
sc_2 = self.f_k(h_mi, c_x)
if s_bias1 is not None:
sc_1 += s_bias1
if s_bias2 is not None:
sc_2 += s_bias2
logits = torch.cat((sc_1, sc_2), 1)
return logits分辨函数和上一篇博客中DGI一致,即s*w*z的模式,这里计算了负样本的全局特征和正样本的全局特征,分别和正样本负样本的局部特征进行了相似性的计算。
所以下一步自然而然的就出来了,计算损失函数。
损失函数包括三部分①重构的基因表达数据的损失,这里计算的是MSE。②正样本的全局特征与正样本的局部特征相似性与1的距离,加上负样本的局部特征的相似性与0的距离。这里用的交叉熵作为损失函数,具体原因在DGI论文中有介绍。③则是②中的距离反过来,②③损失函数说着有点绕,但是逻辑上并不麻烦。三个损失函数加权相加即最终的损失函数。
这里还加了一个正则化损失函数,具体作用稍后会说。
self.loss_sl_1 = self.loss_CSL(ret, self.label_CSL)
self.loss_sl_2 = self.loss_CSL(ret_a, self.label_CSL)
self.loss_feat = F.mse_loss(self.features, self.emb)
if self.add_regularization:
self.loss_norm = 0
for name, parameters in self.model.named_parameters():
if name in ['weight1', 'weight2']:
self.loss_norm = self.loss_norm + torch.norm(parameters, p=2)
loss = self.alpha*self.loss_feat + self.beta*(self.loss_sl_1 + self.loss_sl_2) + self.theta*self.loss_norm
else:
loss = self.alpha*self.loss_feat + self.beta*(self.loss_sl_1 + self.loss_sl_2)这样总的模型部分就全部结束了,简单来说就是在传统的编码器中引入了DGI,做了一个特征提取器。
adata = self.adata_output.copy()
#preprocess(adata)
get_feature(adata)
model = Train(adata, device=self.device)
emb = model.train()
self.adata_output.obsm['emb'] = emb
fix_seed(self.random_seed)
adata = self.adata_output.copy()
#preprocess(adata)
get_feature(adata)
model = Train(adata, add_regularization=True, device=self.device)
emb_regularization = model.train()
self.adata_output.obsm['emb_reg'] = emb_regularization这里通过损失函数是否正则化,训练了两次,模型并得到了两个编码的特征数据。
下一步就是聚类分析。
pca = PCA(n_components=20, random_state=42)
if datatype == '10X' and sample=='single':
# clustering 1
embedding = pca.fit_transform(adata.obsm['emb'].copy())
adata.obsm['emb_pca'] = embedding
adata = mclust_R(adata, used_obsm='emb_pca', num_cluster=n_clusters)
adata.obs['label'] = adata.obs['mclust']
new_type = refine_label(adata, radius, key='label')
adata.obs['label_refined'] = new_type
# clustering 2
embedding = pca.fit_transform(adata.obsm['emb_reg'].copy())
adata.obsm['emb_reg_pca'] = embedding
adata = mclust_R(adata, used_obsm='emb_reg_pca', num_cluster=n_clusters)
adata.obs['label_reg'] = adata.obs['mclust']
new_type = refine_label(adata, radius, key='label_reg')
adata.obs['label_reg_refined'] = new_type分别将编码特征进行PCA降维到20,然后用mclust进行聚类。然后将聚类结果修正一下。
def refine_label(adata, radius=50, key='label'):
n_neigh = radius
new_type = []
old_type = adata.obs[key].values
#read distance
if 'distance_matrix' not in adata.obsm.keys():
raise ValueError("Distance matrix is not existed!")
distance = adata.obsm['distance_matrix'].copy()
n_cell = distance.shape[0]
for i in range(n_cell):
vec = distance[i, :]
index = vec.argsort()
neigh_type = []
for j in range(1, n_neigh+1):
neigh_type.append(old_type[index[j]])
max_type = max(neigh_type, key=neigh_type.count)
new_type.append(max_type)
new_type = [str(i) for i in list(new_type)]
#adata.obs['label_refined'] = np.array(new_type)
return new_type这里的修正函数相当于强制修正,即每个spot半径小于50的范围内,其他spot 的大部分是哪一类就把这个spot 强制归为这一类。这个最初在spaGCN中见过这个方法,SEDR也用了类似的方法好像论文中没有写,所以具体的可以参考spaGCN那篇论文。
这样就有了四个聚类结果,普通编码,正则化后的编码,普通编码修正、正则化编码修正,10x数据一般是要修正后的数据,所以下一步就是在普通编码修正和正则化编码修正中挑选一个结果作为最终结果。
# Silhouette
SIL = metrics.silhouette_score(adata.obsm['emb_pca'], adata.obs['label'], metric='euclidean')
SIL_reg = metrics.silhouette_score(adata.obsm['emb_reg_pca'], adata.obs['label_reg'], metric='euclidean')
if abs(SIL-SIL_reg) > threshold and SIL_reg > SIL:
if refinement:
adata.obs['domain'] = adata.obs['label_reg_refined']
else:
adata.obs['domain'] = adata.obs['label_reg']
else:
if refinement:
adata.obs['domain'] = adata.obs['label_refined']
else:
adata.obs['domain'] = adata.obs['label']挑选方法则是计算编码特征与预测的标签之间的轮廓系数,修正后的轮廓系数比原始的修正系数大,且不是大的太多的时候,选择修正,否则不修正。这里不是太多取的0.06,有大神路过帮忙解释下为什么这么选择。
这样计算一下ari就可以了,这里试过,结果确实非常的好,ari均值在0.55以上。
这样所有的程序就都结束了,解卷积和后续的分析我再学习学习,下一篇博客见。
文章出处登录后可见!
