最近在准备数学建模,凭借微弱的matlab基础学习遗传算法等一系列最优化算法实在是有点吃力。想着帮助自己消化吸收+帮助其他小白同学快速看懂代码,所以写了一篇blog,给出了遗传算法的大致实现思路。但重点是里面傻瓜式的代码注释,详细到了每一个系统函数、matlab语法的用法。适合仓促备赛、快速上手matlab实现算法的同学。
分割线—2023/4/24更新
经验与教训之谈:
这篇文章是本人大一零经验入门数模,学习算法思路时写的文章,当然当时打国赛也用上了这一篇文章里的代码。(对的,当时一切算法代码都是手搓,现在想来有种刺激爽快而很傻的矛盾快感)
但是后来逐渐发现,数模核心其实并不在于编写算法代码—这无异于从零造别人造过的轮子,吃力还低效。学习数模的核心应该是:了解更多可行的算法,学会如何调用/结合现有的最优的代码去解决自己的问题。
因此从这个角度,我不建议数模跑数据时直接用我下面的代码去跑,而可以去调现成的很方便的库。(但是学习时看看底层代码会有更深刻的认知,比赛时附录可以把源码贴上去充实文章)
这篇文章应该是提供一份资料让大家学习了解这个算法,并且在论文里能把这个算法说清楚。
目录
一、遗传算法的思想来源
二、遗传算法的原理分析
①建立种群的基因库——二进制编码
1)确定基因序列长度N
2)建立自变量到十进制数间一一映射关系
②实现遗传过程的交配、突变、选择遗传等过程
1)依适应度的概率选择规则
2)新种群复制
3)新种群交配(交叉)
4)基因突变
5)遗传迭代
二、完整代码段
1)各种参数初始化(需要自己补充上下界、精度)
2)主函数
3)结果可视化(main)
4)适应度计算函数( 自己写目标函数targetfun() )
5)选择复制函数
6)交叉函数
7)变异函数
7)适应度计算函数
8)轮盘赌
9)二进制-十进制转换函数
一、遗传算法的思想来源
遗传算法是建立在自然选择学说基础之上的智能算法。生物种群在自然选择中优胜劣汰,代代繁衍。每一次种群更替,总是淘汰劣质基因、保留优质基因,从而朝着种群的最优发展方向进化。
生物遗传是生物种群不断靠近全局最优解的过程,那么我们能不能用这个思路来解决最优化问题呢?
当然可以:仿照自然遗传过程,①人为设定一个数据组作为“种群基因”,②以目标函数作为评判“发展方向优劣”的标准;③让这群数据通过计算机模拟的“交配”、“基因突变”、“种群复制”等自然选择过程,不断迭代更新、优胜劣汰;④基因趋于稳定时,我们称其为“成熟”,就得出问题的全局最优解。
二、遗传算法的原理分析
①建立种群的基因库——二进制编码
实际的生物基因是由ACTG四个碱基组成的字母串,要模拟生物基因,使用二进制代码01序列是方便的:
1.基因的交叉——01序列部分交换
2.基因突变——二进制数串的某一位0-1翻转
3.二进制与题目求的十进制数之间转换方便
而matlab也可以方便地创建出一个01的矩阵,以模仿生物种群中不同个体的基因序列:
population = round(rand(popsize,Length));
%rand(popsize,length)为随机数函数,生成一个在[0,1]区间的popsize*length大小的随机数矩阵
%round函数对rand函数生成的矩阵四舍五入处理,即可生成01矩阵
%参数说明:population 种群基因库 popsize 种群中个体的大小
生成的population(种群)类似下表:
| 基因序列(长度为length)/ 个体序号(大小为popsize) | |||||||||
| 个体① | 1 | 0 | 0 | 1 | 1 | 0 | 1 | 1 | 0 |
| 个体② | 0 | 1 | 1 | 0 | 1 | 1 | 0 | 0 | 1 |
| …… |
(表1)
01表示的二进制序列很好的解决了基因模拟问题,但现在又有两个新问题:
① Q:如果我们要求某个函数在(-5,5)上的最优解,种群就应该落在这个区间上。但二进制编码到十进制编码都是在0到2^N上的数,如何将其映射到自变量区间上?
② Q:01序列的长度如何选取?
A:由你所要求的自变量精度precision决定。自变量十进制要求精度越高,二进制精度要求当然也要高,基因序列也就越长。
这两个问题都是通过二进制编码实现的,下面通过一个具体题目来感受下这个编码思路:
例:求解下述复杂函数的最大值,自变量x∈[-2,2],要求精度precision=0.001
编码过程:
1)确定基因序列长度N
精度为precision=0.001,那么就将[-2,2]这个区间分成了 4÷0.001=4000个数。按照上面的思路,其中每一个数都应该对应二进制基因序列的一种排列方式。大概如下:
| 自变量 | 二进制(N位) |
| -2.000 | 00……00 |
| -2.001 | 00……01 |
| …… | …… |
| 1.999 | 11……10 |
| 2.000 | 11……11 |
(表2)
所以,二进制最大值 == 编码数量
解得 基因长度N
普遍的,二进制长度公式为
2)建立自变量到十进制数间一一映射关系
二进制序列确定好了之后,对应的十进制数也就确定了
| 二进制 | 十进制 | 自变量 |
| 000…000 | 0 | -2.000 |
| 000…001 | 1 | -1.999 |
| …… | ……. | …… |
| 111…110 | 3999 | 1.999 |
| 111…111 | 4000 | 2.000 |
二进制 —-> 十进制 —->自变量 之间的一一映射关系实际是简单的
二进制到十进制:
function x=transform2to10(population);
%函数功能:将二进制数转换为十进制数
%传入参数:种群的二进制序列population(按行表示)
%返回值:x 种群对应的十进制数
Length = size(population,2);%获取列数即染色体长度
x=population(Length); %x初始化为该染色体的最末位
for i =1:Length-1
x=x+population(Length-i)*power(2,i);
end十进制数转换成自变量区间内的数:
x = lower_bound + x_10 *(upper_bound-lower_bound) / (power(2,Length)-1);
%将十进制数x_10 转化为[lower_bound,upper_bound]区间的自变量x 用等分法
到此,基因序列编码、基因与自变量间的一一对应关系建立完成,就可以使用这群01基因序列进行运算求解实际问题了。
②实现遗传过程的交配、突变、选择遗传等过程
1)依适应度的概率选择规则
生物繁衍过程,种群的生老病死,基因是否突变,基因是否顺利遗传,哪两个基因发生交配等问题都是不确定的概率问题。因此,我们不能用绝对的标准控制某个基因的遗传情况。
因此,我们只能通过抽奖的方式,将某个个体被抽到的概率与其生存能力(适应度)挂钩。生存能力越强,适应度越大,自然也就有更大机会被选中,进行遗传行为了。
生物繁衍过程的适应度是指:生物生存技能、生物体质是否壮硕等指标。类似的,求解函数的最大值问题时,我们自然将自变量的函数值作为适应度了。函数值越大、它适应度越大,越容易被抽中。
同理,当求解最小值优化问题时,应构造适应度函数,使得函数值越小,适应度越大。
具体适应度与概率的评估过程如下:
a.根据目标函数,计算出适应度fitness;对适应度累加,求出种群的总适应度fitness_sum;
求解个体占总适应度的比例,将每个个体按比例分配到0-1区间上的一个小区间。
个体 1 2 3 4 区间 [0,0.1] [0.1,0.4] [0.4,0.9] [0.9,1.0] 占总适应度比例 10 30 50 10 b.由于rand函数生成0-1区间上的数的几率是一定的。因此某个体适应度越大,随机数落在该个体所在区间上的概率就越大。因此使用rand()函数生成在[0,1]区间上的数,进行随机抽奖。
例如,生成随机数若为0.6,那么就落在了个体3所在区间上,个体3被选中。
%子函数fitnessfun() ,计算适应度及累计概率的函数
%返回值fitvalue为适应度,cumsump为概率区间
%population为种群的基因矩阵
function [Fitvalue,cumsump]=fitnessfun(population);
global Length
global lower_bound
global upper_bound
%size函数 输入参数1为获取矩阵的行数(种群大小),2为获取列数(基因长度)
popsize =size(population,1);
%进入循环遍历每个染色体
for i=1:popsize
x_10=transform2to10(population(i,:)); %二进制转换为十进制
%转化为[-2,2]区间的自变量x 用等分法
x=lower_bound+ x_10 *(upper_bound-lower_bound)/(power(2,Length)-1);
Fitvalue(i)=targetfun(x); %求解适应度 targetfun()为目标函数
end
Fitvalue=Fitvalue'+230; %避免出现负适应度,在这里加一个正数
fsum=sum(Fitvalue); %求解总适应度
Pperpopulation = Fitvalue/fsum; %求解每个个体的概率P
cumsump(1)=Pperpopulation(1); %依概率在0,1上分配区间
for i=2:popsize
cumsump(i)=cumsump(i-1)+Pperpopulation(i);
end
cumsump=cumsump'; //转置一下,返回概率区间
抽奖规则做好了,接下来只需要完善基因交叉、遗传、变异等功能函数,组装起来,遗传算法的架构就做好了。
2)新种群复制
种群复制: 该步骤主要为选择一定数量的染色体复制到下一代,是实现“优胜劣汰”的关键步骤。每个个体是否能被遗传到下一代,由上述提到的个体适应度决定。
但是,这里存在两种复制方案,我认为有必要在这里说明一下:
① 将适应度从大到小进行排序,选取适应度最优的P_0个个体遗传到下一代
②依照适应度大小,得出被遗传的概率大小,依照概率随机抽选个体遗传。该方法中,最优个体有可能在复制中被丢失;最差个体也有可能被复制到下一代。
部分小伙伴可能理所当然认为①是最合理的。但并不是,两种方法有其各自的应用范围。下面分别谈谈:
1、办法①在求解一些简单的优化问题,通常局部最优即为全局最优解的问题是适用的。但是,每一步都只保留优秀个体而损失不良个体,可能会被“暂时的优秀”,即局部最优所蒙蔽。这种做法在面对复杂函数时,会失去一定的全局搜索能力。
比如,当函数呈现多峰分布且梯度较大时,种群在某时候聚集在局部最优解附近,这种做法只保留了位于局部最优解附近的种群,而损失了在其他领域探索的不良个体。但有可能,不良个体才是距离全局最优最近的个体。
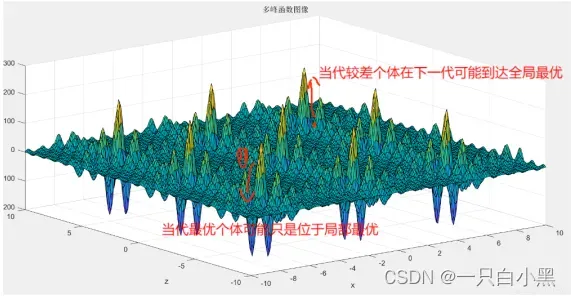
因此,面对复杂问题是,更合理的办法是选择方案二——依概率遗传。这样可以保证对复杂问题的全局搜索性,对解出最终答案至关重要。
function seln = selection(population,cumsump,num);
%子函数selection()
%进行新种群选择操作
%输入参数:种群population,累计概率cumsump,要求遗传个体数num
%返回值为被选中的染色体序号seln
pop_size=size(population,1); %获取当前种群规模
selected=zeros(size(population,1));%用于标记每个个体是否被选择过(避免重复)
for i =1:1:num
while 1 %循环包裹,若当前选中了一个未被选过的个体时跳出,否则重新来
r=rand; %产生一个随机数
prand=cumsump-r; %概率区间统一减去随机数。
index=1;
while prand(index)<0 %第一个正数所在区间就是被选中的染色体区间。
index=index+1;
end
if selected(index)~=1 %判断是否被选过
selected(index)=1; %赋值
break;
end
end
seln(i)=index; %选中个体的序号
end
3)新种群交配(交叉)
每次交叉的两个染色体为步骤2)中选出的染色体,将其序号作为参数传入crossover子函数中,是否交配遵循以下规则:
①依交叉概率pcrossover,使用下函数随机决定两个染色体是否发生交叉(后面判断是否变异也用它)。
%变异或交叉的抽奖函数 pcc==1 表示抽中,反之没抽中
function pcc = IfCroIfMut(p_crossover);
n=rand;
if n<=p_crossover
pcc=1;
else
pcc=0;
end
②若发生交叉,再使用rand函数抽取一个交叉节点,将两个染色体节点前、后的部分交叉
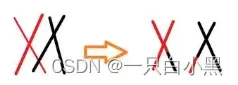
function scro = crossover(population,seln,pcrossover)
%新种群交叉函数:crossover()
%输入参数:原种群,交叉染色体序号,交叉概率
%返回值:交叉后染色体
Length = size(population,2); %取染色体长度为length
pcc = IfCroIfMut(pcrossover); %概率决定是否交叉,1是0否
if pcc==1
position =(round(rand*(Length-2)))+1; %随机选取一个交叉节点
%小白扫盲:矩阵格式(a,b:c,d)---左从a到b行,右从c到d列依次遍历
%取seln(1)的position前序列,seln(2)的position后序列交叉
scro(1,:)=[population(seln(1),1:position) ,population(seln(2),position+1:Length)];
scro(2,:)=[population(seln(2),1:position) ,population(seln(1),position+1:Length)];
else
scro(1,:)=population(seln(1),:);
scro(2,:)=population(seln(2),:);
end4)基因突变
基因变异即将基因序列上的某一位进行0-1翻转。变异的基因为由rand函数随机产生。
是否发生变异由变异概率pmutation决定。
function snew = mutation(population,pmutation);
%突变函数mutation()
%进行变异步骤 population为原种群,pmutation为变异概率
%返回值为snew,承载新种群的染色体信息。
Length = size(population,2); %获取基因长度
%snew为返回值
snew=population;
pmm = IfCroIfMut(pmutation);
if pmm==1
position=round(rand*(Length-1))+1; %抽变异的染色体位置
snew(position)=abs(population(position)-1); %0-1翻转
end5)主函数 遗传迭代
将函数块组合到主函数中,进行遗传迭代
%计算适应度,返回适应度fitvalue 累积概率 cunsump
[Fitvalue, cumsump] = fitnessfun(population);
Generation =1;
while Generation<Generation_max+1
for j=1:2:popsize %j和j+2两两交叉
%选择,返回seln为选择结果
seln = selection(population,cumsump);
%交叉 返回scro为交叉后的两根染色体
scro = crossover (population,seln,p_crossover);
%加入交叉后的染色体 定义scnew为进行遗传步骤后的新种群
scnew(j,:)=scro(1,:);
scnew(j+1,:)=scro(2,:);
%变异 smnew为在scnew基础上变异后的新种群,最新了
smnew(j,:)=mutation(scnew(j,:),p_mutation);
smnew(j+1,:)=mutation(scnew(j+1,:),p_mutation);
end
population = smnew; %产生新种群
%计算新种群适应度
[Fitvalue,cumsump]=fitnessfun(population);
%记录当代最好适应度与平均适应度
[fmax,nmax]=max(Fitvalue);
fmean = mean(Fitvalue);
%ymax记录每一代的最好适应度与平均适应度
ymax(Generation)=fmax;
ymean(Generation)=fmean;
%记录当代最佳染色体个体
x_10=transform2to10(population(nmax,:));
%自变量取【-2,2】,需要把经过遗传运算的最佳染色体整合到该区间。
x=lower_bound+x_10*(upper_bound-lower_bound)/(power((upper_bound),Length)-1);
xmax(Generation)=x;
Generation=Generation+1;
end
Generation=Generation-1; %由于while中generation+1后跳出循环,所以减回去
Bestpopulation=x;
Besttargetfunvalue=targetfun(x);二、优化原理分析——交叉、变异步骤的意义
由上述讨论我们已经清楚,遗传算法实际上是通过模拟自然界优胜劣汰过程,求解最优化问题的算法。但我们还有一个疑问:优胜劣汰的过程其实通过“选择”这一个步骤就可实现,为什么出现了额外的交叉、变异呢?
实际上,交叉与变异分别对应了“全局搜索”与“局部搜索”两种性能。
①交叉,是两个染色体直接进行交换。该步骤使得染色体序列发生较大变动,最终染色体映射到的实数也会发生较大变化,使得种群分散,体现出算法的全局搜索性。
②变异,是但染色体序列中某一位出现0-1翻转。该步骤使得染色体序列在小范围内波动,体现出局部的搜索性。
交叉与变异,分别影响了算法的全局搜索性能和局部搜索性能,那么,我们可以调整交叉与变异的强度,从而使算法更倾向于全局搜索或者局部搜索,以适应不同的优化问题。
比如:
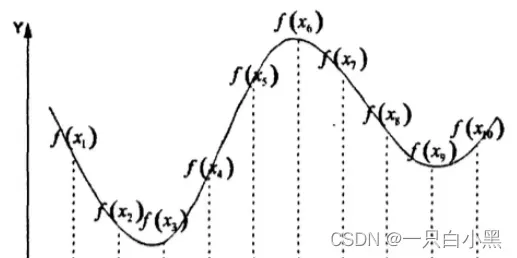
如果优化函数是全局性多峰值函数,那么,全局搜索性能更重要。为避免种群陷入局部最优解,我们可以增大 交叉概率p_crossover。
如果优化函数在最优解附近梯度比较大,需要精细的搜索,那么我们可以通过增大变异概率p_mutation,从而提升算法的局部搜索性能。
三、完整代码段
该程序适用于求解目标函数最大值。如需求解最小值,将主函数中记录最大值部分改为最小值即可。
1)各种参数初始化(需要自己补充上下界、精度)
%各种参数的初始化
global Length %length标记染色体长度
global lower_bound %标记自变量取值区间下界
global upper_bound %自变量取值区间上界
precision = ; %设置取值精度
lower_bound = ; %取下界
upper_bound = ; %取上届
%求解染色体长度。ceil(x)函数,将x向上取整并返回
Length = ceil(log2((upper_bound-lower_bound)'./precision));
popsize = 50; %设置种群大小
Generation_max =12; %迭代次数上限
p_crossover =0.9; %交配概率
p_mutation=0.09; %突变概率
%rand()为随机数函数,随机生成popsize(行,代表每一条染色体) * length(列数,染色体的基因序列)矩阵
%round函数对矩阵每个数四舍五入(以获取01序列的染色体)
population = round(rand(popsize,Length)); %初始化种群2)主函数
%计算适应度,返回适应度fitvalue 累积概率 cunsump
[Fitvalue, cumsump] = fitnessfun(population);
Generation =1;
while Generation<Generation_max+1
for j=1:2:popsize %j和j+2两两交叉
%选择,返回seln为选择结果
seln = selection(population,cumsump);
%交叉 返回scro为交叉后的两根染色体
scro = crossover (population,seln,p_crossover);
%定义scnew为进行遗传步骤后的新种群
scnew(j,:)=scro(1,:);
scnew(j+1,:)=scro(2,:);
%变异 smnew为在scnew基础上变异后的新种群,最新了
smnew(j,:)=mutation(scnew(j,:),p_mutation);
smnew(j+1,:)=mutation(scnew(j+1,:),p_mutation);
end
population = smnew; %产生新种群
%计算新种群适应度
[Fitvalue,cumsump]=fitnessfun(population);
%记录当代最好适应度与平均适应度
[fmax,nmax]=max(Fitvalue);
fmean = mean(Fitvalue);
%ymax记录每一代的最好适应度与平均适应度
ymax(Generation)=fmax;
ymean(Generation)=fmean;
%记录当代最佳染色体个体
x_10=transform2to10(population(nmax,:));
%自变量取【-2,2】,需要把经过遗传运算的最佳染色体整合到该区间。
x=lower_bound+x_10*(upper_bound-lower_bound)/(power((upper_bound),Length)-1);
xmax(Generation)=x;
Generation=Generation+1;
end
Generation=Generation-1; %由于while中generation+1后跳出循环,所以减回去
Bestpopulation=x;
Besttargetfunvalue=targetfun(x);3)结果可视化(main)
%绘制运算后的适应度曲线。
%若平均适应度与最大适应度在曲线上有趋同态势,表示算法收敛较顺利,没出现震荡。此前提下,若最大适应度个体连续若干代无发生变化,表明种群成熟
figure(1); %创建序号1图窗
hand1=plot(1:Generation,ymax);
set(hand1,'linestyle','-','linewidth',1.8,'marker','*','markersize',6)
hold on;
hand2 = plot(1:Generation,ymean);
set(hand2,'color','r','linestyle','-','linewidth',1.8,'marker','h','markersize',6);
xlabel('进化代数');ylabel('最大/平均适应度');xlim([1 Generation_max]);
legend('最大适应度','平均适应度');
box off;hold off;4)适应度计算函数( 自己写目标函数targetfun() )
%计算适应度函数fitnessfun ,返回值fitvalue为适应度,cumsump为累计概率
function [Fitvalue,cumsump]=fitnessfun(population);
global Length
global lower_bound
global upper_bound
%size函数,获取population的大小,输入参数1位获取行数,2为获取列数
popsize =size(population,1);
%进入循环遍历每个染色体
for i=1:popsize
x_10=transform2to10(population(i,:));%取种群的第i行,求解其十进制
%转化为[-2,2]区间的实数xx 用等分法
x=lower_bound+x_10*(upper_bound-lower_bound)/(power(upper_bound,Length)-1);
%求解适应度 targetfun为目标函数
Fitvalue(i)=targetfun(x);
end
%'运算符为转置
%这里+230的目的是:在不改变适应度相对大小的前提下,将其全部转化为正数,以将适应度归一化。230为经验参数,可改
Fitvalue=Fitvalue'+230;
%求解总适应度
fsum=sum(Fitvalue);
Pperpopulation = Fitvalue/fsum; %适应度0-1化
%计算累积概率
cumsump(1)=Pperpopulation(1);
for i=2:popsize
cumsump(i)=cumsump(i-1)+Pperpopulation(i);
end
cumsump=cumsump';5)选择复制函数
%子函数selection()
%新种群选择操作:每次选中两个染色体
%返回值为被选中的染色体序号seln
function seln = selection(population,cumsump);
%从种群选择两个个体
for i =1:2
r=rand; %产生一个随机数
prand=cumsump-r; %概率区间统一减去随机数。
j=1;
while prand(j)<0 %第一个正数所在区间就是被选中的染色体区间。
j=j+1;
end
seln(i)=j; %选中个体的序号
end
6)交叉函数
%新种群交叉函数:crossover()
%参数为:原种群,交叉染色体序号,交叉概率
%返回值为交叉后染色体
function scro = crossover(population,seln,pcrossover);
Length = size(population,2); %取染色体长度为length
pcc = IfCroIfMut(pcrossover); %概率决定是否交叉,1是0否
if pcc==1
position =(round(rand*(Length-2)))+1; %随机选取一个交叉节点
%小白扫盲:矩阵格式(a,b:c,d)---左从a到b行,右从c到d列依次遍历
%取seln(1)的position前序列,seln(2)的position后序列交叉
scro(1,:)=[population(seln(1),1:position) ,population(seln(2),position+1:Length)];
scro(2,:)=[population(seln(2),1:position) ,population(seln(1),position+1:Length)];
else
scro(1,:)=population(seln(1),:);
scro(2,:)=population(seln(2),:);
end7)变异函数
%突变函数mutation()
%返回值为snew,承载新种群的染色体信息。
%进行变异步骤 population为原种群,pmutation为变异概率
function snew = mutation(population,pmutation);
Length = size(population,2); %获取基因长度
%snew为返回值
snew=population;
pmm = IfCroIfMut(pmutation);
if pmm==1
position=round(rand*(Length-1))+1; %抽变异的染色体位置
snew(position)=abs(population(position)-1); %0-1翻转
end7)适应度计算函数
%计算适应度函数fitnessfun ,返回值fitvalue为适应度,cumsump为累计概率
function [Fitvalue,cumsump]=fitnessfun(population);
global Length
global lower_bound
global upper_bound
%size函数,获取population的大小,输入参数1位获取行数,2为获取列数
popsize =size(population,1);
%进入循环遍历每个染色体
for i=1:popsize
x_10=transform2to10(population(i,:));%取种群的第i行,求解其十进制
%转化为[-2,2]区间的实数xx 用等分法
x=lower_bound+x_10*(upper_bound-lower_bound)/(power(upper_bound,Length)-1);
%求解适应度 targetfun为目标函数
Fitvalue(i)=targetfun(x);
end
%'运算符为转置
Fitvalue=Fitvalue'+230;
%求解总适应度
fsum=sum(Fitvalue);
Pperpopulation = Fitvalue/fsum; %适应度0-1化
%计算累积概率
cumsump(1)=Pperpopulation(1);
for i=2:popsize
cumsump(i)=cumsump(i-1)+Pperpopulation(i);
end
cumsump=cumsump';8)轮盘赌
%变异或交叉的抽奖函数
function pcc = IfCroIfMut(pcrossover);
test(1:100)=0; %建立1x100的全零矩阵
L = round(100*pcrossover); %取0-100p内随机数
test(1:L)=1; %从1到l都变为1 ,建立与p成正比的抽奖概率
n=round(rand*99)+1;
pcc=test(n); %抽奖
9)二进制-十进制转换函数
%将二进制数转换为十进制数
function x=transform2to10(population);
%获取列数即染色体长度
Length = size(population,2);
%注意 population为某一染色体的基因序列
x=population(Length); %x初始化为该染色体的最末位
%倒序求解翻译成10进制
for i =1:Length-1
x=x+population(Length-i)*power(2,i);
end
博主水平能力有限,有错误请及时指正!
(如果对你有帮助,也请点个小小的赞)
文章出处登录后可见!
