可视化计算化学的分子
从“微笑”到 3D

化学中的微笑……不,我们不打算谈论上图中的那个。由于实验失败,这些微笑通常很少发生在实验室或计算机模拟中:-)。我们将讨论一些与计算化学有关的东西。简而言之,计算化学是化学的一个分支,它使用计算机工具来协助复杂的化学研究,如药物设计。自从阿尔法折叠和量子计算出现以来,该领域正在经历一场新的复兴。
SMILES(简化分子输入行输入系统)是一种基于行符号中的字符的符号,该符号使用短 ASCII 字符串描述化合物的结构。大多数分子编辑器可以导入 SMILES 字符串并将它们转换回二维图形(尽管分子实际上是 3D 的)。这种方法非常有用,因为它允许您利用基于字符串操作和自然语言处理的最新机器学习工具。下图显示了从化学结构编码到 SMILES 表示的几个示例(例如 CC(=)….):
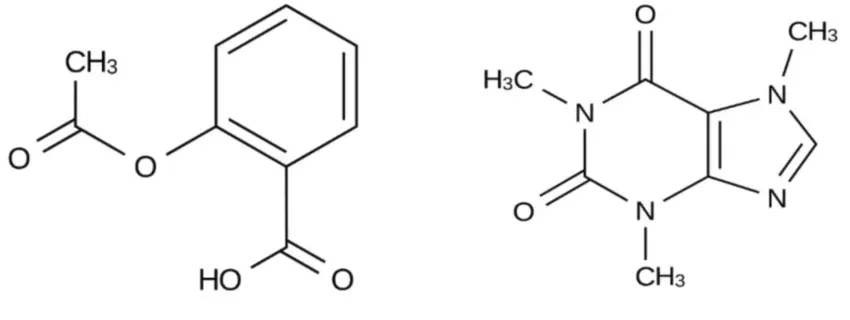
如前所述,大多数编辑器(如 ChemDraw)将 SMILES 符号转换为 2D 绘图,即使分子实际上是 3D 的。幸运的是,字符中的信息允许使用更高级的工具来重建完整的 3D 表示,这就是本文的主题。
在大多数 3D 文件格式中,有 .pdb(蛋白质数据库文件)或更简单的 .mol 。鉴于它与 RDKit 等 Python 库和 PyMol 等免费工具的多功能性,我们在此描述 .mol 文件格式。让我们首先探索如何将 SMILES 符号转换为 .mol 文件格式,然后是 .mol 文件中的内容,以及使用 PyMol 可视化的最终结果。[0][1][2]
下面的代码片段从 RDKIT 包中导入一些库,将它们绘制成上图中的 2D a,最终将分子保存到 .mol 文件中。在 CoLab 上运行此代码的笔记本可在此处获得。[0][1]
关键是读取 SMILES 符号的函数“Chem.MolFromSmiles()”。
from rdkit import Chemfrom rdkit.Chem import AllChemfrom rdkit.Chem import Drawfrom rdkit.Chem.Draw import IPythonConsole
# Convert SMILE into .molmy_mol=Chem.MolFromSmiles('NC(=N)N1CCC[C@H]1Cc2onc(n2)c3ccc(Nc4nc(cs4)c5ccc(Br)cc5)cc3')my_mol_with_H=Chem.AddHs(my_mol)AllChem.EmbedMolecule(my_mol_with_H)AllChem.MMFFOptimizeMolecule(my_mol_with_H)my_embedded_mol = Chem.RemoveHs(my_mol_with_H)
#Save the molecule into an imageDraw.MolToFile(my_mol,'molecule.png')#Save molecular rapresentation in mol filesfout = Chem.SDWriter('./charged_test.mol')fout.write(my_embedded_mol)fout.close()
作为 RDKIT 一个 Python 库,你可以想象它可以很容易地与 Python 中的机器学习、自然语言处理和其他 AI 库相结合。
.mol 文件格式是一个文本 ASCII 文件,以空格分隔的方式包含几个元素,它们是:
- 原子列表,每个原子都指定了其元素身份。
- 指定它们连接的原子的键列表以及键的多重性(单,双,三)
- 每个原子的 2D 或 3D 空间坐标
- 计算分子中原子和键的数量。
- 与原子或键相关的属性
- 与整个结构相关的属性。
这些通常以列或块的形式表示,如下图所示。第一列是原子的 3D 坐标,然后是原子符号,然后是属性,最后是键值。如果您想了解更多有关此结构的详细信息,请查看 Robert Belford 的“MOL 文件解剖”。[0]

通过使用 RDKIT 的代码获得的 .mol 可以使用 PyMol 可视化为:
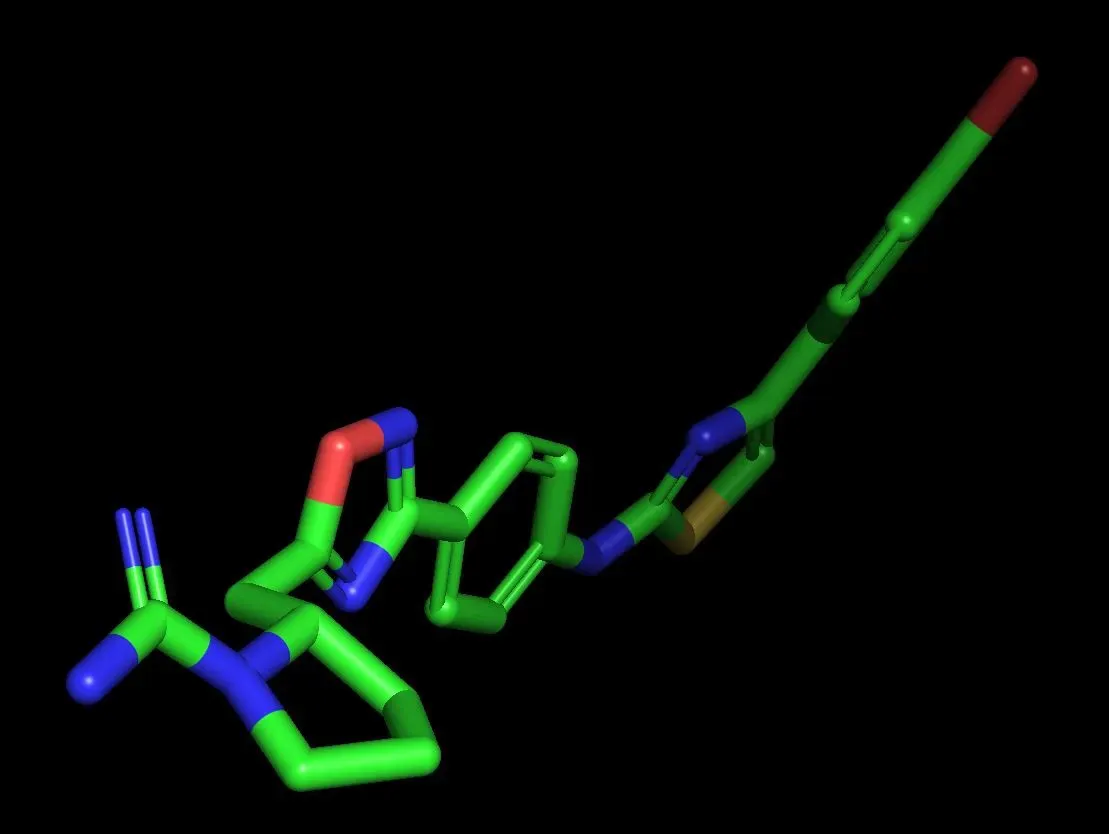
总之,我们在这里介绍了一些在 Python 脚本和可视化工具中从 SMILES 表示法恢复分子结构 3D 的简单方法。这是为了证明使用 SMILES 符号仍然很方便,即使它看起来不那么可读。尽管在如何在 3D 空间中定向分子方面出现了许多问题,但药物的图形表示也可以用于机器学习的上下文中。 SMILES 符号尽管对人类来说显然很麻烦,但它可以利用我们在机器学习中的自然语言处理已经取得的所有进步,包括最近的许多作品所示。[0]
References
PyMOL | pymol.org[0]
RDKIT[0]
Chemdraw[0]
MOL 文件的剖析[0]
张等人。 Frontiers in Chemistry 2020 “SPVec: A Word2vec-Inspired Feature Representation Method for Drug-Target Interaction Prediction”[0]
PDB文件剖析[0]
文章出处登录后可见!
