遗传算法的具体运用之TSP问题的Python解决之法
文章目录
前言
随着人工智能的不断发展,机器学习这门技术也越来越重要,很多学科都渐渐接触到机器学习的内容,本文章就经典算法——遗传算法做一个简要介绍,并以典型的TSP问题展示遗传算法的运用。本文章的程序使用Python编程语言进行书写。
提示:以下是本篇文章正文内容,下面案例可供参考
一、基本概念简述
遗传:子代与父代的某些部分具有相同或相似的形状,以保证物种的稳定性。
变异:子代与父代、子代不同个体之间存在差异性。
进化:生物在演化过程中,物种逐渐适应环境,淘汰了不适应环境的物种,留下了能够适应环境的优良物种,这种现象称为进化。
二、算法简介
2.1 遗传算法介绍
遗传算法是1975年由密切根大学的J.Holland提出的,它是一种通过模拟自然进化过程寻找最优解的方法。例如,可以利用它求解函数的最值问题。
2.2 基因与染色体
在遗传算法中,我们首先需要将一个现实问题转化为具体的函数表达式,我们称着一过程为“数学建模”,该问题的一个可行解称为一条“染色体”
例如:对于不等式x + y + z < 60, [1, 2, 3], [5, 7, 9], [8, 2, 5]都是这个不等式函数的可行解,每一个可行解在遗传算法中被称为染色体,每一个可行解中的每一个元素称为染色体上的一个基因。
2.3 个体编码
遗传算法的运算对象是表示格提的符号串,需要对“数学建模”后的变量x,y,z编码为一种符号串。常见的编码方式为使用无符号二进制数来表示。
2.4 初始群体的产生
遗传算法是对群体进行的进化操作,需要准备一些表示起始搜索点的起始群体数据。
例如,群体的规模大小为4,即群体由4个个体组成,每个个体可以通过随机方法生成。例如:011, 010, 100, 110。
2.5 遗传算法中的物竞天择
2.5.1“物竞”(适应度函数)
能够适应环境的概率==>适应度函数遗传算法中以个体适应度的大小来评定各个个体的优劣程度,从而决定其遗传机会的大小。
2.5.2 “天择”(选择函数)
在自然界中,越适应的个体就越有可能繁殖后代。但也不是适应度越高就肯定后代越多,只能是从概率上来说是更多。例如,日常生活中可能见到的转盘游戏。
2.6 交叉与变异
交叉:
父: 00110101
母: 01110001
子: 00110001
变异:
父: 00110101
母: 01110001
子: 10110001
复制:每次进化中,为了保留上一代优良的染色体,需要将上一代中适应度最高的几条染色体直接原封不动地复制给下一代。
三、应用问题描述
1.TSP问题简述
TSP,(全称 Travelling salesman problem)。旅行商客问题主要解决的是最佳路径规划问题。针对这一类典型问题,常用的优化算法蚁群算法、模拟退火算法、遗传算法等等。本文采用遗传算法进行优化。
2. 数据集介绍
TSP问题根据问题的不同所需要的数据集也不同,为降低难度进行理解,本次实验采用的是二维数据集,即通过两个数值表示一个城市点的坐标。提升一点难度可以考虑三维模式,再难一点就可以考虑球坐标点,这样就需要读者通过坐标转化关系式进行建模。针对这几种数据集,无论是哪一种,最终在使用遗传算法时都是十分类似的,通常都是使用距离作为评判标准和优化目标。
本次所采用的数据集部分截图展示如下:

3. 问题重述
本次实验所要解决的问题是对TSP数据进行处理,构建合适的遗传算法程序和评判指标,寻找出最优的路径。
四、算法编程实现
1. 导入第三方库
本次实验主要涉及到数据处理与可视化第三方库numpy和matplotlib,导入第三方库具体程序如下:
import numpy as np
import matplotlib.pyplot as plt
2. 准备数据集
2.1 测试数据集
本次实验首先随机生成10个地址,对编写的程序做检验,具体程序如下:
class TSP(object):
def __init__(self, citys):
data = np.random.rand(citys, 2)
self.city_position = data
2.2 TSP问题数据处理与导入
常规TSP问题数据集导入与预处理
data = np.loadtxt("data12_1.txt")
# 将导入的数据集转换成(x,y)的形式
data1 = np.array(data).reshape(-1, 2)
通过TSP类导入数据
class TSP(object):
def __init__(self, data1):
self.city_position = data1
env = TSP(data1 = data1)
在TSP类程序里,除了定义了数据集,同时也定义了图像绘制程序,可视化的显示数据处理的过程。具体程序展示如下:
class TSP(object):
def plotting(self, lx, ly, total_d):
plt.cla()
plt.scatter(self.city_position[:, 0].T, self.city_position[:, 1].T, s=100, c='k')
plt.plot(lx.T, ly.T, 'r-')
plt.text(-0.05, -0.1, "Total distance=%.2f"%total_d, fontdict={'size': 20, 'color': 'red'})
# plt.xlim(0.1, 1.1)
# plt.ylim(-0.15, 1.1)
plt.pause(0.01)
3. 遗传算法程序书写
class GA(object):
def __init__(self, citys, pc, pm, popsize):
self.citys = citys
self.pc = pc
self.pm = pm
self.popsize = popsize
# 生成种群,DNA序列为0到50的随机序列
self.pop = np.vstack([np.random.permutation(citys) for _ in range(popsize)])
# 将种群中排列好的序列横纵坐标分别提取出来
def translateDNA(self, DNA, city_position):
lineX = np.empty_like(DNA, dtype=np.float64)
lineY = np.empty_like(DNA, dtype=np.float64)
for i, d in enumerate(DNA):
city_coord = city_position[d]
lineX[i, :] = city_coord[:, 0]
lineY[i, :] = city_coord[:, 1]
return lineX, lineY
# 求取距离适应度函数
def getFitness(self, lineX, lineY):
totalDis = np.empty((lineX.shape[0],), dtype=np.float64)
for i, (xval, yval) in enumerate(zip(lineX, lineY)):
totalDis[i] = np.sum(np.sqrt(np.square(np.diff(xval)) + np.square(np.diff(yval))))
fitness = np.exp(self.citys * 2 / totalDis)
return fitness, totalDis
# 定义选择函数
def selection(self, fitness):
idx = np.random.choice(np.arange(self.popsize), size=self.popsize, replace=True, p=fitness/fitness.sum())
return self.pop[idx]
# 交叉函数
def crossover(self, parent, pop):
if np.random.rand() < self.pc:
i = np.random.randint(0, self.citys, size=1)
cross_points =np.random.randint(0, 2, self.citys).astype(np.bool)
keep_city = parent[~cross_points]
swap_city = pop[i, np.isin(pop[i].ravel(), keep_city, invert=True)]
parent[:] = np.concatenate((keep_city, swap_city))
return parent
# 变异函数
def mutation(self, child):
for point in range(self.citys):
if np.random.rand() < self.pm:
swap_point = np.random.randint(0, self.citys)
swapa, swapb = child[point], child[swap_point]
child[point], child[swap_point] = swapa, swapb
return child
# 进化函数
def evolve(self, fitness):
pop = self.selection(fitness)
pop_copy = pop.copy()
for parent in pop:
child = self.crossover(parent, pop_copy)
child = self.mutation(child)
parent[:] = child
self.pop = pop
4. 结果展示
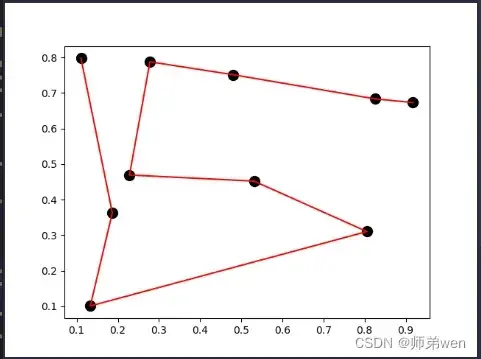
测试程序结果展示如下:
TSP问题展示结果如下:
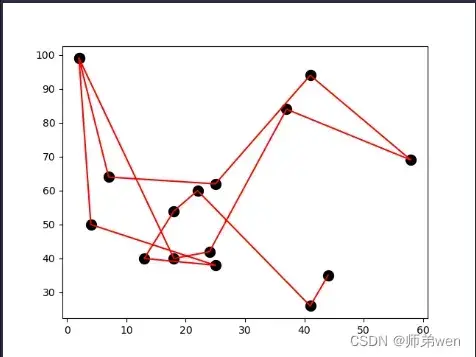
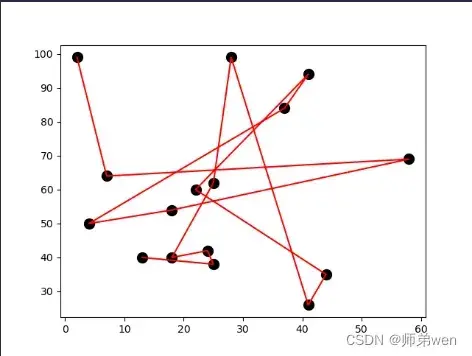
5. 附录
测试程序:
import numpy as np
import matplotlib.pyplot as plt
# data = np.loadtxt("data12_1.txt")
# data1 = np.array(data).reshape(-1, 2)
citys = 10
pc = 0.23
pm = 0.037
popsize = 50
iternum = 200
class GA(object):
def __init__(self, citys, pc, pm, popsize):
self.citys = citys
self.pc = pc
self.pm = pm
self.popsize = popsize
# 生成种群,DNA序列为0到50的随机序列
self.pop = np.vstack([np.random.permutation(citys) for _ in range(popsize)])
# 将种群中排列好的序列横纵坐标分别提取出来
def translateDNA(self, DNA, city_position):
lineX = np.empty_like(DNA, dtype=np.float64)
lineY = np.empty_like(DNA, dtype=np.float64)
for i, d in enumerate(DNA):
city_coord = city_position[d]
lineX[i, :] = city_coord[:, 0]
lineY[i, :] = city_coord[:, 1]
return lineX, lineY
# 求取距离适应度函数
def getFitness(self, lineX, lineY):
totalDis = np.empty((lineX.shape[0],), dtype=np.float64)
for i, (xval, yval) in enumerate(zip(lineX, lineY)):
totalDis[i] = np.sum(np.sqrt(np.square(np.diff(xval)) + np.square(np.diff(yval))))
fitness = np.exp(self.citys * 2 / totalDis)
return fitness, totalDis
# 定义选择函数
def selection(self, fitness):
idx = np.random.choice(np.arange(self.popsize), size=self.popsize, replace=True, p=fitness/fitness.sum())
return self.pop[idx]
# 交叉函数
def crossover(self, parent, pop):
if np.random.rand() < self.pc:
i = np.random.randint(0, self.citys, size=1)
cross_points =np.random.randint(0, 2, self.citys).astype(np.bool)
keep_city = parent[~cross_points]
swap_city = pop[i, np.isin(pop[i].ravel(), keep_city, invert=True)]
parent[:] = np.concatenate((keep_city, swap_city))
return parent
# 变异函数
def mutation(self, child):
for point in range(self.citys):
if np.random.rand() < self.pm:
swap_point = np.random.randint(0, self.citys)
swapa, swapb = child[point], child[swap_point]
child[point], child[swap_point] = swapa, swapb
return child
# 进化函数
def evolve(self, fitness):
pop = self.selection(fitness)
pop_copy = pop.copy()
for parent in pop:
child = self.crossover(parent, pop_copy)
child = self.mutation(child)
parent[:] = child
self.pop = pop
class TSP(object):
def __init__(self, citys):
# self.data = np.loadtxt("data12_1.txt")
# self.data1 = np.array(self.data).reshape(-1, 2)
# self.x = []
# self.y = []
# for i in range(citys):
# self.x.append(self.data1[i][0])
# self.y.append(self.data1[i][1])
# data = [[3, 2],
# [6, 8],
# [9, 5],
# [10, 20],
# [18, 13],
# [6, 9],
# [9, 11],
# [3, 5],
# [7, 9],
# [8, 3]]
data = np.random.rand(citys, 2)
self.city_position = data
plt.ion()
def plotting(self, lx, ly, total_d):
plt.cla()
plt.scatter(self.city_position[:, 0].T, self.city_position[:, 1].T, s=100, c='k')
plt.plot(lx.T, ly.T, 'r-')
plt.text(-0.05, -0.1, "Total distance=%.2f"%total_d, fontdict={'size': 20, 'color': 'red'})
# plt.xlim(0.1, 1.1)
# plt.ylim(-0.15, 1.1)
plt.pause(0.01)
if __name__ == "__main__":
env = TSP(citys=citys)
ga = GA(citys=citys, pc=pc, pm=pm, popsize=popsize)
for gen in range(iternum):
lx, ly = ga.translateDNA(ga.pop, env.city_position)
fitness, total_distance = ga.getFitness(lx, ly)
ga.evolve(fitness)
best_idx = np.argmax(fitness)
print("Gen:", gen, "| best fit: %.2f"%fitness[best_idx],)
env.plotting(lx[best_idx], ly[best_idx], total_distance[best_idx])
plt.ioff()
plt.show()
实验程序:
# 导入第三方库
import numpy as np
import matplotlib.pyplot as plt
data = np.loadtxt("data12_1.txt")
data1 = np.array(data).reshape(-1, 2)
citys = len(data1)
pc = 0.39
pm = 0.01
popsize = 20
iternum = 2000
class GA(object):
def __init__(self, citys, pc, pm, popsize):
self.citys = citys
self.pc = pc
self.pm = pm
self.popsize = popsize
# 生成种群,DNA序列为0到50的随机序列
self.pop = np.vstack([np.random.permutation(citys) for _ in range(popsize)])
# 将种群中排列好的序列横纵坐标分别提取出来
def translateDNA(self, DNA, city_position):
lineX = np.empty_like(DNA, dtype=np.float64)
lineY = np.empty_like(DNA, dtype=np.float64)
for i, d in enumerate(DNA):
city_coord = city_position[d]
lineX[i, :] = city_coord[:, 0]
lineY[i, :] = city_coord[:, 1]
return lineX, lineY
# 求取距离适应度函数
def getFitness(self, lineX, lineY):
totalDis = np.empty((lineX.shape[0],), dtype=np.float64)
for i, (xval, yval) in enumerate(zip(lineX, lineY)):
totalDis[i] = np.sum(np.sqrt(np.square(np.diff(xval)) + np.square(np.diff(yval))))
fitness = np.exp(self.citys * 2 / totalDis)
return fitness, totalDis
# 定义选择函数
def selection(self, fitness):
idx = np.random.choice(np.arange(self.popsize), size=self.popsize, replace=True, p=fitness/fitness.sum())
return self.pop[idx]
# 交叉函数
def crossover(self, parent, pop):
if np.random.rand() < self.pc:
i = np.random.randint(0, self.citys, size=1)
cross_points =np.random.randint(0, 2, self.citys).astype(np.bool)
keep_city = parent[~cross_points]
swap_city = pop[i, np.isin(pop[i].ravel(), keep_city, invert=True)]
parent[:] = np.concatenate((keep_city, swap_city))
return parent
# 变异函数
def mutation(self, child):
for point in range(self.citys):
if np.random.rand() < self.pm:
swap_point = np.random.randint(0, self.citys)
swapa, swapb = child[point], child[swap_point]
child[point], child[swap_point] = swapa, swapb
return child
# 进化函数
def evolve(self, fitness):
pop = self.selection(fitness)
pop_copy = pop.copy()
for parent in pop:
child = self.crossover(parent, pop_copy)
child = self.mutation(child)
parent[:] = child
self.pop = pop
class TSP(object):
def __init__(self, data1):
# self.data = np.loadtxt("data12_1.txt")
# self.data1 = np.array(self.data).reshape(-1, 2)
# self.x = []
# self.y = []
# for i in range(citys):
# self.x.append(self.data1[i][0])
# self.y.append(self.data1[i][1])
self.city_position = data1
plt.ion()
def plotting(self, lx, ly, total_d):
plt.cla()
plt.scatter(self.city_position[:, 0].T, self.city_position[:, 1].T, s=100, c='k')
plt.plot(lx.T, ly.T, 'r-')
plt.text(-0.05, -0.1, "Total distance=%.2f"%total_d, fontdict={'size': 20, 'color': 'red'})
# plt.xlim(0.1, 1.1)
# plt.ylim(-0.15, 1.1)
plt.pause(0.01)
if __name__ == "__main__":
env = TSP(data1=data1)
ga = GA(citys=citys, pc=pc, pm=pm, popsize=popsize)
for gen in range(iternum):
lx, ly = ga.translateDNA(ga.pop, env.city_position)
fitness, total_distance = ga.getFitness(lx, ly)
ga.evolve(fitness)
best_idx = np.argmax(fitness)
print("Gen:", gen, "| best fit: %.2f"%fitness[best_idx],)
env.plotting(lx[best_idx], ly[best_idx], total_distance[best_idx])
plt.ioff()
plt.show()
提示:注释部分的程序为调试时所用,读者复现时可以做一定的参考。
总结
本次实验基本利用遗传算法解决了TSP问题,但是在实验中也发现了很多不足之处。
首先,交叉和变异值的选取没有合理的定义规范,对于后期应对复杂问题具有一定的挑战性。
其次,由于遗传算法存在随机性,最优结果的确定是一个概率问题,由于随机性的存在,导致程序运行中可能已经存在了最优解,但是程序没有中断,可能会导致最后的结果次优,或更差。
最后,将数据不断扩大可以发现,程序的运行效率降低许多的同时寻找到的结果离最优解也差得越来越多。
针对以上存在的问题,也欢迎读者见谅并提出指导性建议,共同探讨和完善。
Ps:该文章由本人和另一博主共同完成。
文章出处登录后可见!