文章目录
- 前言
- 一、导入相关库
- 二、加载Cora数据集
- 三、定义DeepWalk
- 四、可视化
- 完整代码
前言
大家好,我是阿光。
本专栏整理了《图神经网络代码实战》,内包含了不同图神经网络的相关代码实现(PyG以及自实现),理论与实践相结合,如GCN、GAT、GraphSAGE等经典图网络,每一个代码实例都附带有完整的代码。
正在更新中~ ✨

🚨 我的项目环境:
- 平台:Windows10
- 语言环境:python3.7
- 编译器:PyCharm
- PyTorch版本:1.11.0
- PyG版本:2.1.0
💥 项目专栏:【图神经网络代码实战目录】
本文我们将使用Pytorch + Pytorch Geometric来简易实现一个DeepWalk,让新手可以理解如何PyG来搭建一个简易的图网络实例demo。
一、导入相关库
本项目我们需要结合两个库,一个是Pytorch,因为还需要按照torch的网络搭建模型进行书写,第二个是PyG,因为在torch中并没有关于图网络层的定义,所以需要torch_geometric这个库来定义一些图层。
import matplotlib.pyplot as plt
import torch
from sklearn.manifold import TSNE
from torch_geometric.datasets import Planetoid
from torch_geometric.nn import Node2Vec
二、加载Cora数据集
本文使用的数据集是比较经典的Cora数据集,它是一个根据科学论文之间相互引用关系而构建的Graph数据集合,论文分为7类,共2708篇。
- Genetic_Algorithms
- Neural_Networks
- Probabilistic_Methods
- Reinforcement_Learning
- Rule_Learning
- Theory
这个数据集是一个用于图节点分类的任务,数据集中只有一张图,这张图中含有2708个节点,10556条边,每个节点的特征维度为1433。
# 1.加载Cora数据集
dataset = Planetoid(root='./data/Cora', name='Cora')
本项目是使用 Node2Vec 来生成每个节点的特征,所以对于原始节点特征是无用的,本项目只是单纯利用 Cora 数据集的节点空间关系,也就是 edge_index ,基于节点的空间关系来生成对应的节点特征,最终验证生成的节点特征效果如何。
三、定义DeepWalk
这里我们就不重点介绍DeepWalk了,相信大家能够掌握基本原理,本文我们使用的是PyG定义这个网络,在PyG中已经定义好了 Node2Vec 这个层,我们可以利用这个层来实现 DeepWalk 。
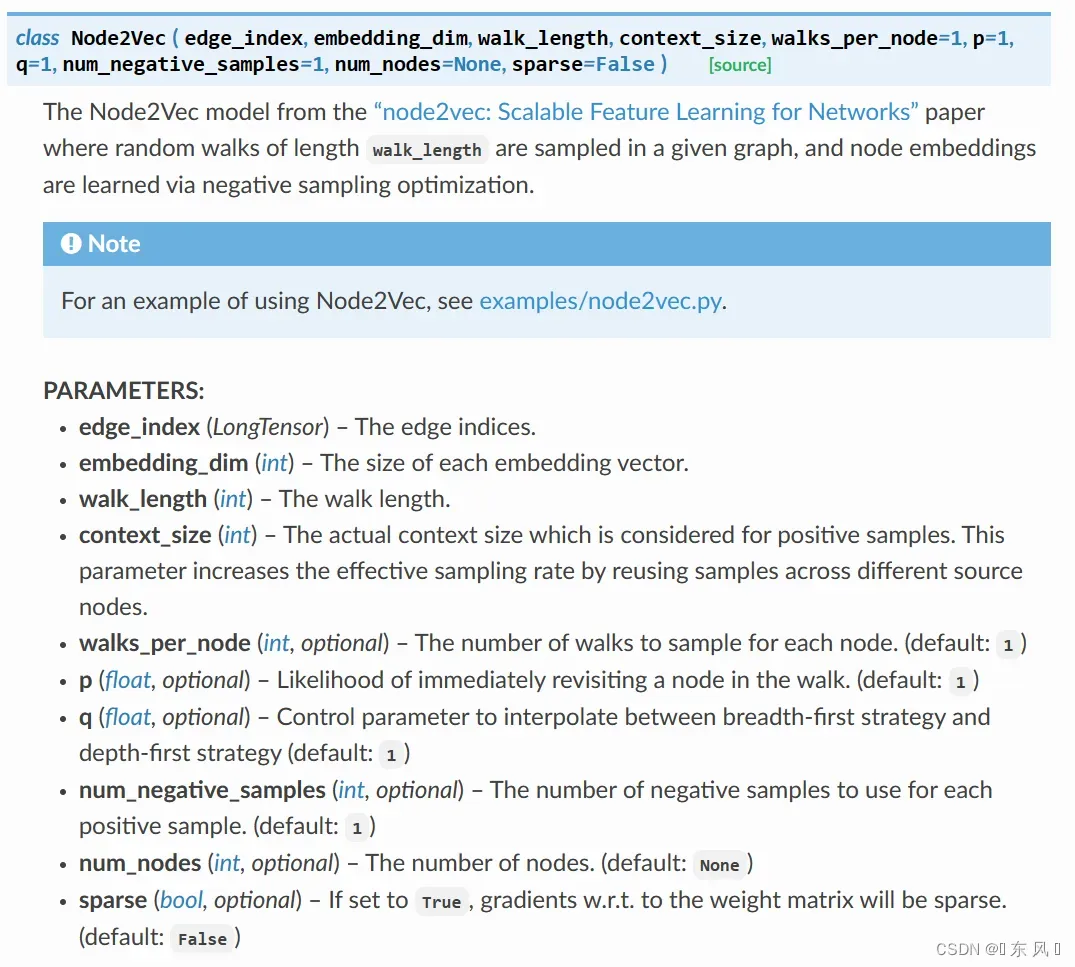
对于Node2Vec的常用参数:
- edge_index:图数据中的边关系,就是
data的edge_index,形状为【2,num_edges】 - embedding_dim:每个节点形成的嵌入维度
- walk_length:游走形成的序列长度
- context_size:上下文大小
- walks_per_node:每个节点形成多少个游走序列
- p:在游走时重新访问某个节点的概率,默认为1
- q:在BFS策略和DFS策略之间的控制参数,默认为1
- num_negative_samples:每个正样本对应的负样本数,默认为1
- num_nodes:图的节点数目
- sparse:如果设置为True,权重矩阵的梯度是以稀疏矩阵方式存储,默认为False
如果熟悉 DeepWalk 和 Node2Vec 两个算法的小伙伴可以发现,如果把 Node2Vec 在游走时设置的概率 p 和 q 同时设为1,此时 Node2Vec 就会退化成为 DeepWalk 。
# deepwalk模型
model = Node2Vec(edge_index=data.edge_index,
embedding_dim=128, # 节点维度嵌入长度
walk_length=5, # 序列游走长度
context_size=4, # 上下文大小
walks_per_node=1, # 每个节点游走10个序列
p=1,
q=1,
sparse=True # 权重设置为稀疏矩阵
).to(device)
对于模型训练等部分,与 Node2Vec 实现方式一致,所以这里不再赘述,如果不清楚的小伙伴可以先去查看本传内的这篇文章 PyG基于Node2Vec实现节点分类及其可视化,这篇文章详细介绍了代码实战部分。
四、可视化
上面我们以经训练好了 DeepWalk 这个模型,通过调用 model() 即可获得内部的权重矩阵,也就是我们要的Embedding向量表(lookup table)。
生成好每个节点的 Embedding,我们可以通过可视化的方式更加直观的看到效果如何,对于可视化操作我们利用的是 TSNE 这个模块来进行降维,因为绘制二维图形需要x轴和y轴坐标(即二维),降到两个维度后,就获得了每个节点的坐标信息,然后利用 matplotlib 这个库来绘制不同类别的节点信息。
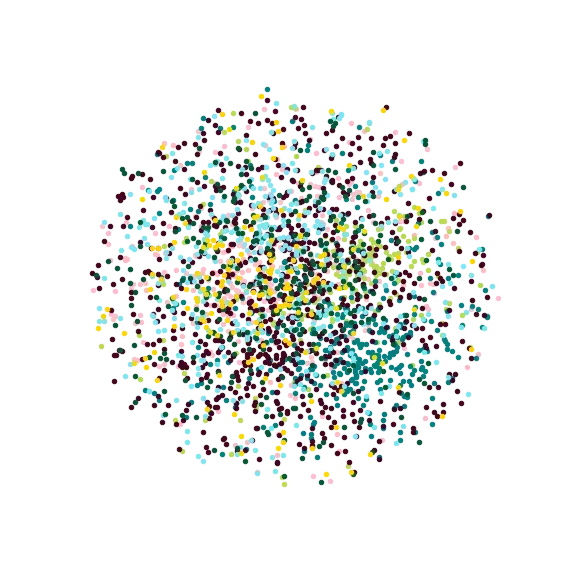
# 可视化节点的embedding
with torch.no_grad():
# 不同类别节点对应的颜色信息
colors = [
'#ffc0cb', '#bada55', '#008080', '#420420', '#7fe5f0', '#065535',
'#ffd700'
]
model.eval() # 开启测试模式
# 获取节点的embedding向量,形状为[num_nodes, embedding_dim]
z = model(torch.arange(data.num_nodes, device=device))
# 使用TSNE先进行数据降维,形状为[num_nodes, 2]
z = TSNE(n_components=2).fit_transform(z.detach().numpy())
y = data.y.detach().numpy()
plt.figure(figsize=(8, 8))
# 绘制不同类别的节点
for i in range(dataset.num_classes):
# z[y==0, 0] 和 z[y==0, 1] 分别代表第一个类的节点的x轴和y轴的坐标
plt.scatter(z[y == i, 0], z[y == i, 1], s=20, color=colors[i])
plt.axis('off')
plt.show()
完整代码
import matplotlib.pyplot as plt
import torch
from sklearn.manifold import TSNE
from torch_geometric.datasets import Planetoid
from torch_geometric.nn import Node2Vec
# 1.加载Cora数据集
dataset = Planetoid(root='../data/Cora', name='Cora')
data = dataset[0]
# 2.定义模型
device = torch.device('cuda' if torch.cuda.is_available() else 'cpu') # 设备
# deepwalk模型
model = Node2Vec(edge_index=data.edge_index,
embedding_dim=128, # 节点维度嵌入长度
walk_length=5, # 序列游走长度
context_size=4, # 上下文大小
walks_per_node=1, # 每个节点游走1个序列
p=1,
q=1,
sparse=True # 权重设置为稀疏矩阵
).to(device)
# 迭代器
loader = model.loader(batch_size=128, shuffle=True)
# 优化器
optimizer = torch.optim.SparseAdam(model.parameters(), lr=0.01)
# 3.开始训练
model.train()
for epoch in range(1, 101):
total_loss = 0 # 每个epoch的总损失
for pos_rw, neg_rw in loader:
optimizer.zero_grad()
loss = model.loss(pos_rw.to(device), neg_rw.to(device)) # 计算损失
loss.backward()
optimizer.step()
total_loss += loss.item()
# 使用逻辑回归任务进行测试生成的embedding效果
with torch.no_grad():
model.eval() # 开启测试模式
z = model() # 获取权重系数,也就是embedding向量表
# z[data.train_mask] 获取训练集节点的embedding向量
acc = model.test(z[data.train_mask], data.y[data.train_mask],
z[data.test_mask], data.y[data.test_mask],
max_iter=150) # 内部使用LogisticRegression进行分类测试
# 打印指标
print(f'Epoch: {epoch:02d}, Loss: {total_loss:.4f}, Acc: {acc:.4f}')
# 可视化节点的embedding
with torch.no_grad():
# 不同类别节点对应的颜色信息
colors = [
'#ffc0cb', '#bada55', '#008080', '#420420', '#7fe5f0', '#065535',
'#ffd700'
]
model.eval() # 开启测试模式
# 获取节点的embedding向量,形状为[num_nodes, embedding_dim]
z = model(torch.arange(data.num_nodes, device=device))
# 使用TSNE先进行数据降维,形状为[num_nodes, 2]
z = TSNE(n_components=2).fit_transform(z.detach().numpy())
y = data.y.detach().numpy()
plt.figure(figsize=(8, 8))
# 绘制不同类别的节点
for i in range(dataset.num_classes):
# z[y==0, 0] 和 z[y==0, 1] 分别代表第一个类的节点的x轴和y轴的坐标
plt.scatter(z[y == i, 0], z[y == i, 1], s=20, color=colors[i])
plt.axis('off')
plt.show()
文章出处登录后可见!
