一、原始数据处理
如图:
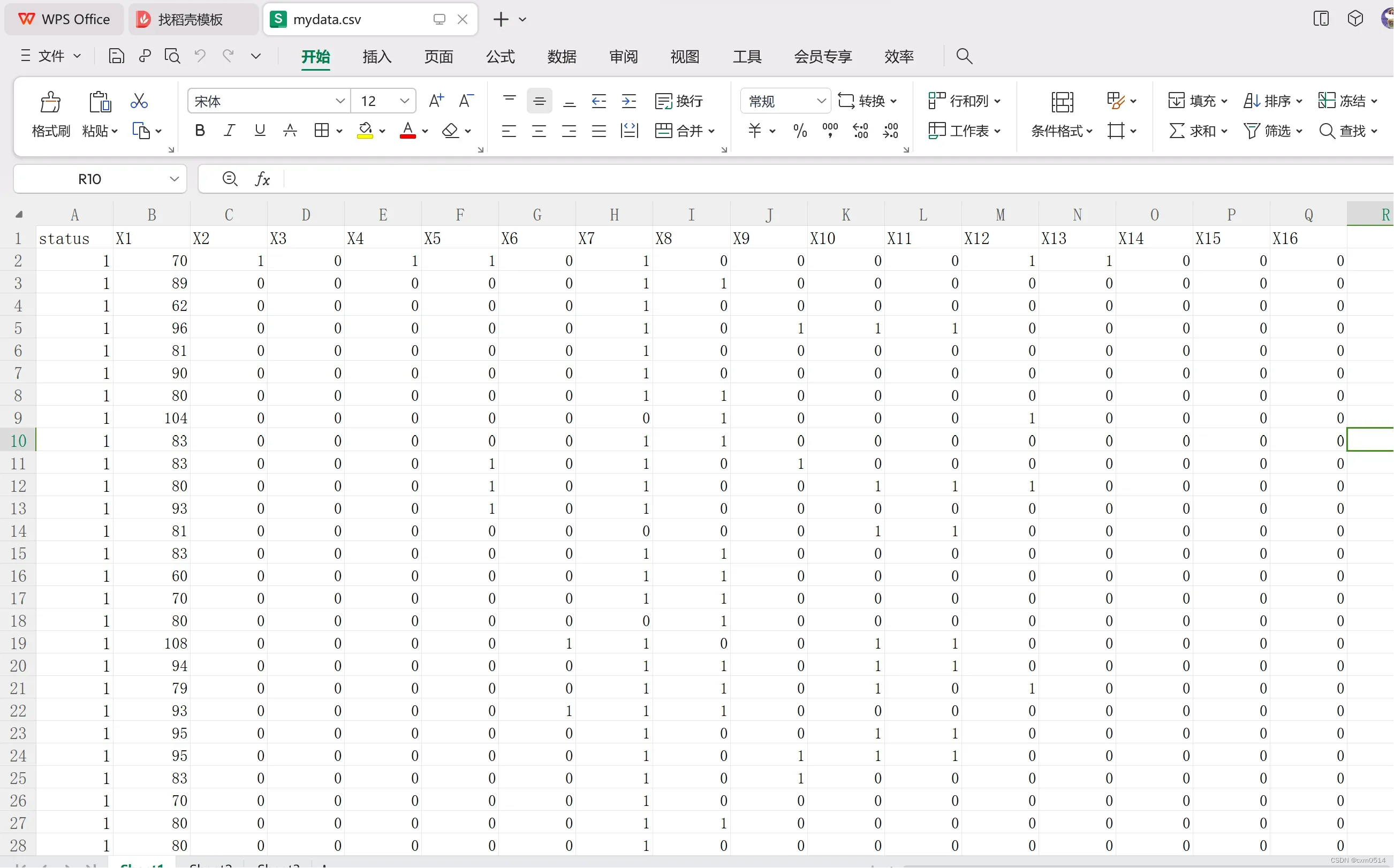
结局status为二分类变量(用0,1表示)
自变量为X1~X15
数据文件名为mydata.csv
二、将数据导入Rstudio
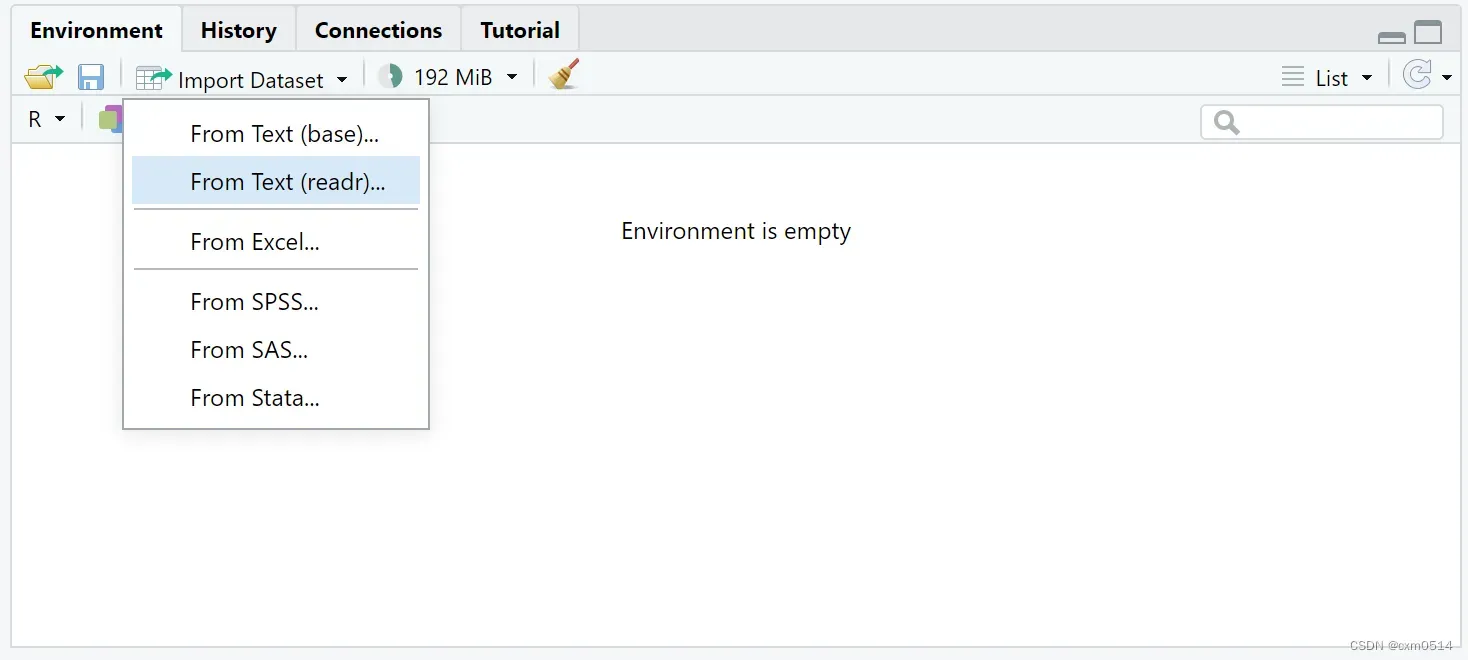 点readr后点击browse找到你的数据,点击Import就可以导入进来了。
点readr后点击browse找到你的数据,点击Import就可以导入进来了。
三、R代码进行LASSO回归
library(glmnet)#加载glmnet包
colnames(mydata[,1:17])#查看前17列的列名(根据自己数据调整)
y <- as.matrix(mydata[, 1]) # 提取第1列作为结局(建议放在第一列)
x <- as.matrix(mydata[, 2:17]) # 第2至第17列为自变量
#后边的代码除了s值基本不需更改
lasso_model <- glmnet(x, y, family = "binomial",
alpha = 1) # 表示采用L1正则化,即Lasso回归。
max(lasso_model$lambda)
print(lasso_model)
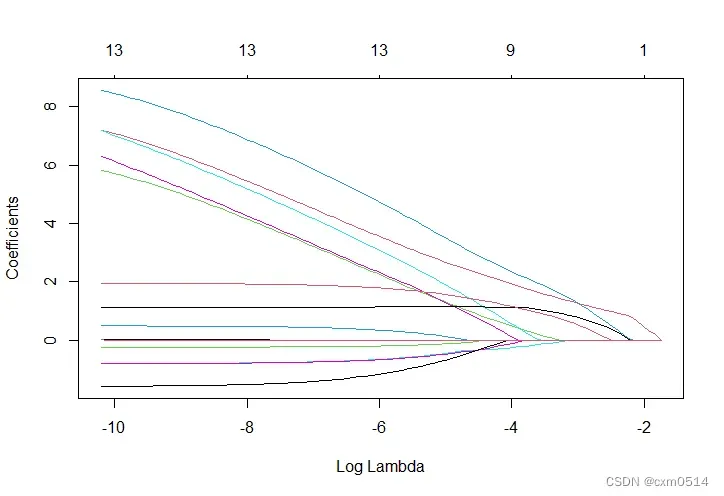
#绘制LASSO图
plot(lasso_model,
xvar = "lambda")
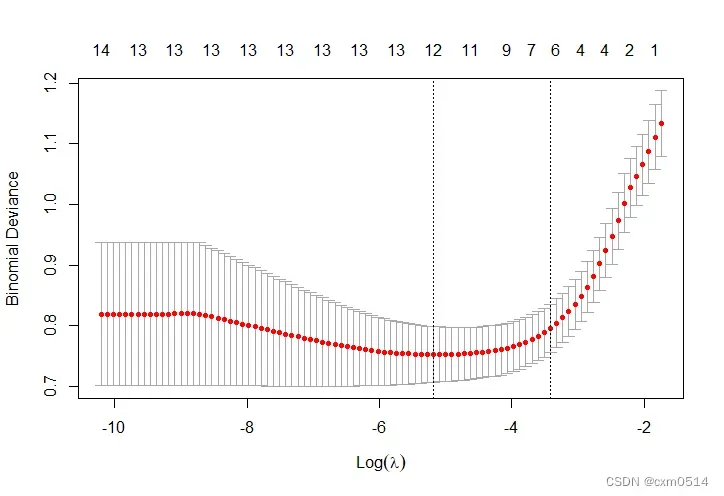
#交叉验证并绘制可视化结果
cv_model <- cv.glmnet(x, y, family = "binomial",alpha = 1,nfolds = 10)
plot(cv_model)
#根据交叉验证结果,选择lambda值,lambda.min或lambda.1se。
lambda_min <- cv_model$lambda.min
lambda_min
lambda_1se <- cv_model$lambda.1se
lambda_1se
#s为Lambda大小,Lambda越大表示模型的正则化强度越大,选择的自变量也越少。
#这里选择的是刚刚得到的lambda_1se的值
coef_lasso <- coef(lasso_model,
s = 0.03955501)
coef_lasso
#结果显示后边带有数值的变量为筛选得到的变量如下图所示,第一张图为plot(lasso_model,xvar=“lambda”)的结果
第二张图为plot(cv_model)的结果
文章出处登录后可见!
已经登录?立即刷新
