作者单位:耶路撒冷希伯来大学
发表期刊:《Bioinformatics》,2020年期刊影响因子:6.937
发表时间:2022年1月9日
数据和代码:https://github.com/nadavbra/protein_bert
1. 研究背景
提出了一个基于自监督的ProteinBERT一个专门为蛋白质设计的深度语言模型。我们的预训练方案将语言建模与基因本体论(GO)注释预测结合起来。引入了新颖的建筑元素,使模型高效且灵活地适应长序列。ProteinBERT的体系结构由局部和全局表示组成,允许对这些类型的输入和输出进行端到端处理。ProteinBERT在涵盖多种蛋白质特性(包括蛋白质结构、翻译后修饰和生物物理属性)的多个基准上获得了接近最先进的性能,具有速度快,拟合速度好的优点。
2. 研究数据
2.1 预训练的蛋白质数据集
ProteinBERT在106M蛋白上进行了预训练UniProtKB/UniRef90,UniRef90提供了一组非冗余的蛋白质簇,至少共享90%的序列一致性。每个簇由一个具有代表性的蛋白质表示,确保蛋白质空间的相对均匀覆盖。对于每个蛋白质,提取其氨基酸序列和相关的氧化石墨烯注释(根据UniProtKB)。我们只考虑了在UniRef90中出现至少100次的8943个最频繁的GO注释。在106M的UniRef90蛋白中,46M至少有8943个被考虑的注释中的一个(在46M蛋白中平均每个蛋白有2.3个注释)。
2.2 蛋白质基准数据集
在9个基准数据集上对其进行了测试,包括蛋白质的功能、结构、翻译后修饰和生物物理特性(如下表所示)。这些基准中的标签要么是局部的(如翻译后修饰),要么是全局的(如远程同源性),它们要么是连续的(如蛋白质稳定性),要么是二元的(如信号肽),要么是分类的(如二级结构)。在局部基准中,训练样本的数量远远大于蛋白质序列的数量,因为目标标签是每个残基。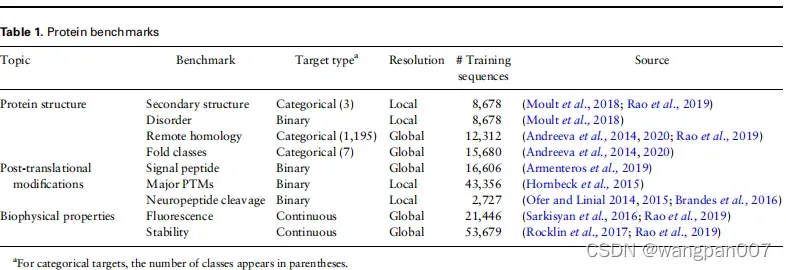
3. 研究方法
3.1 序列和标注编码
蛋白质序列被编码成整数记号序列。我们使用26个标记来代表20个标准氨基酸,硒半胱氨酸(U),一个未定义的氨基酸(X),另一个氨基酸(OTHER)和3个额外标记(START, END和PAD)。对于每个序列,分别在第一个氨基酸之前和最后一个氨基酸之后添加START和END标记。将PAD令牌添加到短于小批处理所选序列长度的PAD序列中。
ProteinBERT的体系结构(像大多数深度学习模型一样)规定了每个小批都有固定的序列长度。我们包括了START和END标记,以帮助模型解释长于所选序列长度的蛋白质。当编码一个超过所选序列长度的蛋白质时,我们选择该蛋白质的一个随机子序列,至少去掉它的两端之一。START或END令牌的缺失允许模型识别到它只接收到序列的一部分。
每个序列的GO注释被编码为固定大小的二进制向量(8943),其中除与该蛋白相关的GO注释对应的条目外,所有条目均为零。当没有向模型提供GO注释的信息时(例如:
在基准的调整和评估期间),向量将全部设置为零。
3.2 蛋白质序列和注释的自我监督预训练
对蛋白质序列和从UniRef90中提取的GO注释进行了预处理。模型接收到损坏的输入(蛋白质序列和氧化石墨烯注释),必须恢复未损坏的数据。蛋白质序列的破坏采用5%概率随机替换令牌的方式进行(即保持原始令牌的95%概率,或用一个均匀选择的5%概率随机替换令牌)。对输入的GO注释进行破坏,随机去除现有的注释,概率为25%,并添加随机错误的注释,每个与蛋白质不相关的注释的概率为0.01%。对于50%的加工蛋白质,我们将所有的输入注释全部删除(即给出一个全零输入向量),以迫使模型仅从序列预测氧化石墨烯注释(就像所有测试基准的情况一样)。总之,所描述的预训练是一项双重任务,其中模型必须恢复蛋白质序列及其已知的GO注释。由于氧化石墨烯术语涵盖了广泛的功能,后一项任务与蛋白质研究的许多领域相关。
3.3 对蛋白质基准进行监督微调
对于所有基准,ProteinBERT都从相同的预训练状态初始化,并通过相同的协议进行微调。最初,预训练模型的所有层都被冻结,只有新添加的完全连接的层被允许训练40个epoch。接下来,我们解冻所有的图层,并训练模型达到40个额外的时代。最后,我们为一个更大序列长度的最后epoch训练模型,在所有时期,我们在平台上降低了学习速率,并基于独立的验证集应用了早期停止。模型评估,然后在一个保留的测试集上执行。在整个调优和基准测试评估过程中,没有利用GO注释的信息(即GO注释输入始终是一个常量的全零向量)。在单个GPU上,整个微调过程花费了14分钟。
3.4 深度学习框架
roteinBERT的架构是不同的,包括几个创新。ProteinBERT是一种去噪自动编码器(下图所示)。ProteinBERT的两个输入(和输出)分别是(i)蛋白质序列(编码为氨基酸标记序列)和(ii) GO注释(编码为固定大小的二进制向量)。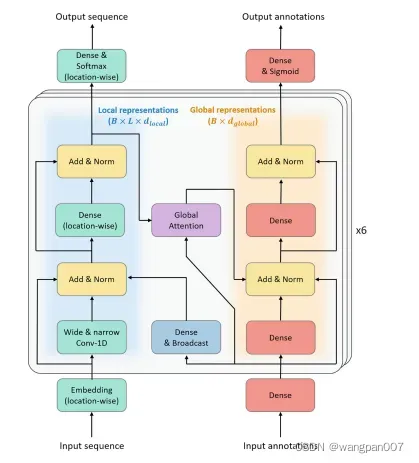
局部和全局表示由六个类似变压器的块处理,这些块具有跳过连接和隐藏层之间的层标准化。在每个块内,局部表示首先通过一维卷积层进行转换,然后通过一个(基于位置的)全连接层进行转换。为了允许每个位置的局部表示在短距离和远距离上都基于其他位置,我们使用了窄卷积层(无膨胀)和宽卷积层(膨胀率为5)。两种类型的卷积层的核大小都是9 。
本地和全局表示之间的唯一信息流是通过广播全连接层(从全局到本地表示)和全局注意层(从本地到全局表示)发生的。广播层是完全连接的层,它们将全局表示的dglobal特征转换为局部表示的dlocal特征,然后在每个L序列位置上复制该表示。
受自注意机制的启发,全球注意层具有线性(而不是二次)复杂性。self-attention时需要输入序列和输出另一个序列通过允许每个职位参加彼此的位置,全球关注的需要作为输入序列和一个全球固定大小固定大小的向量和输出向量由出席的每一个地方输入职位根据全球输入向量。具有参数量少的特点,仅仅包括16M的训练参数。
在标记数据集上微调ProteinBERT时,另一层被添加到其输出中。最后一层提供模型的局部或全局隐藏状态的连接,这取决于输出标签是局部的还是全局的。最后一层使用的激活取决于输出类型(例如,分类标签的softmax激活,二进制标签的sigmoid激活,连续标签的不激活)。
4. 结果
4.1 预训练可以改善蛋白质模型
在106M UniRef90记录上进行了6.4个epoch的预训练,改变用于编码输入和输出蛋白序列的序列长度(128、512或1024个记号)。我们观察到128令牌编码的性能较低,但512和1024的性能类似。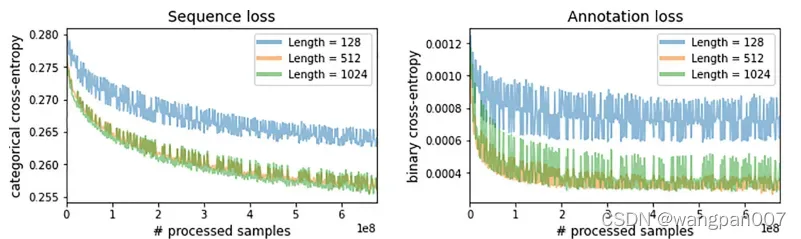
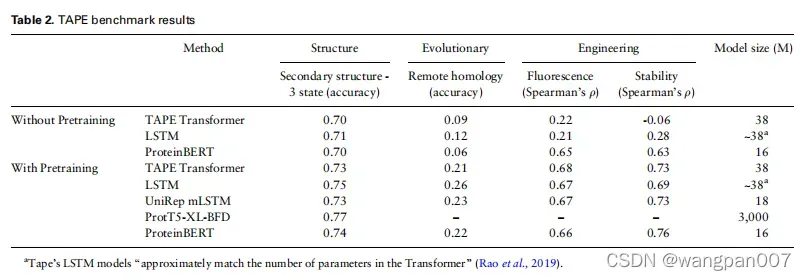
4.2 ProteinBERT在不同的蛋白质基准上达到了近乎最先进的结果
使用了九个涵盖蛋白质研究中各种任务的基准(下表所示)。对于来自TAPE的四个基准(二级结构、远程同源性、荧光和稳定性预测),我们将我们的性能与其他最先进的序列模型进行了比较,这些模型在相同的基准和相同的指标下进行了评估。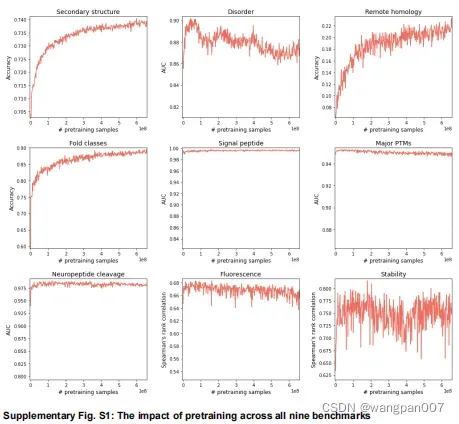
为了进一步了解预训练对下游基准性能的影响,根据不同的预训练时间评估了ProteinBERT。具体来说,我们从预训练过程中的不同快照启动模型,并在从这些状态进行微调后评估其下游性能(图3)。虽然一些任务不能从预训练中受益,但其他任务(如二级结构和远程同源性)显示出从越来越多的预训练中获得的明显收益,并没有在改善中显示出饱和。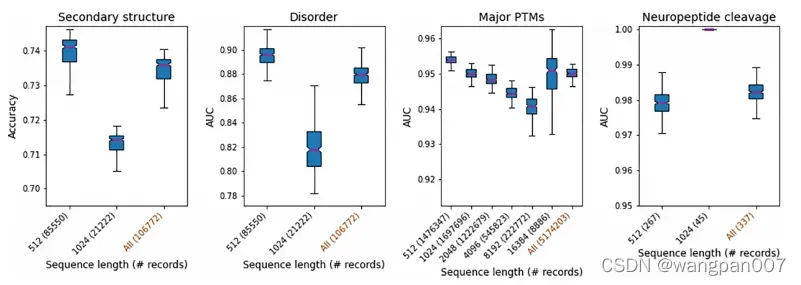 消融试验来研究GO-annotation预训练任务(下图),发现一些基准(特别是二级结构、远程同源性和折叠类)从中受益。测量了ProteinBERT在9个基准中的4个上的测试集性能,这些基准具有不可忽略的数量的测试集记录在超过512令牌的蛋白质(图4)。具体来说,我们需要至少25个这样的记录,其中,一个记录包含一个完整的蛋白质(在全局任务的情况下)或一个残基(在局部任务的情况下)。我们观察到,在大多数情况下,ProteinBERT在较长的序列上表现稍差,但也只是轻微的,这表明它确实适用于很宽范围的蛋白质长度
消融试验来研究GO-annotation预训练任务(下图),发现一些基准(特别是二级结构、远程同源性和折叠类)从中受益。测量了ProteinBERT在9个基准中的4个上的测试集性能,这些基准具有不可忽略的数量的测试集记录在超过512令牌的蛋白质(图4)。具体来说,我们需要至少25个这样的记录,其中,一个记录包含一个完整的蛋白质(在全局任务的情况下)或一个残基(在局部任务的情况下)。我们观察到,在大多数情况下,ProteinBERT在较长的序列上表现稍差,但也只是轻微的,这表明它确实适用于很宽范围的蛋白质长度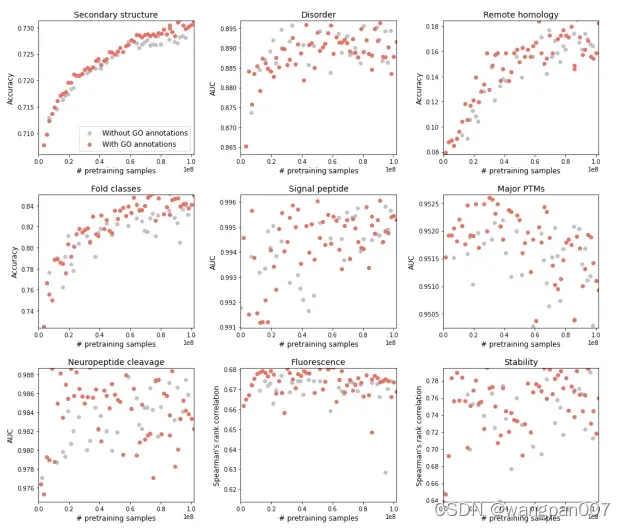
4.4 全局注意力机制的理解
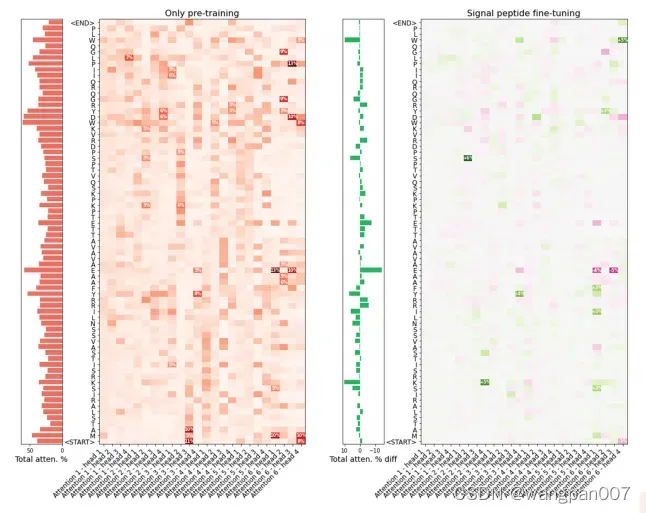
为了证明整体注意机制的内部工作原理,我们从信号肽基准的测试集中选取了两个不相关的蛋白质,提取了蛋白bert中的24个注意头值,在对该任务的模型进行微调之前和之后(图5)。不同蛋白质之间的整体注意力模式明显不同,但也存在一些共同的模式。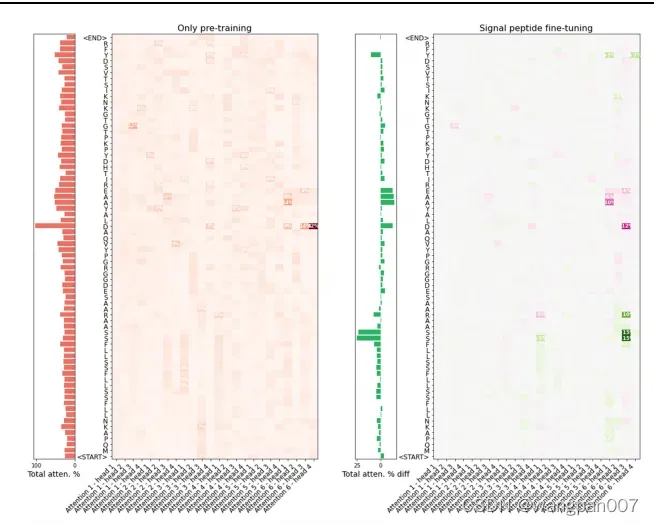
5. 结论
提出了一种新的蛋白质序列深度语言模型ProteinBERT,旨在以一种自然的方式捕获蛋白质的局部和全局表示。我们已经证明了该模型的普遍性。表明它可以在几分钟内对各种各样的蛋白质任务进行微调,并达到接近最先进的结果。尽管一些较大的蛋白质语言模型[如ProtT5 ]在至少一些测量任务上显示出更好的性能,这些模型要大得多,在预训练和推理过程中涉及的计算量和内存都要大几个数量级。
版权声明:本文为博主wangpan007原创文章,版权归属原作者,如果侵权,请联系我们删除!
原文链接:https://blog.csdn.net/wangpan007/article/details/123259292
