B 站地址:https://www.bilibili.com/video/BV1vN411o7r3?p=2[0]
1. DNA 和染色体
1.1. DNA 的化学组成
单核苷酸:磷酸-[磷酸键]-戊糖-[糖苷键]-碱基
- 碱基 base: 嘌呤 (Adenine/Guanine):二元环,有氨基(-NH2);G 有氧嘧啶 (Cytosine/Thymine/Uracil):一元环,C 只有一个氧,有氨基;T 有甲基-CH3核酸的修饰:碱基上的氨基被氧化
- 戊糖 ribose: 戊糖 2,3,5 位有羟基-OH,核糖核苷酸的 2’是羟基,脱氧核糖核苷酸的戊糖 2’无氧(脱氧),3’或 5’羟基与磷酸形成磷酸酯键。DNA 合成方向:5’→3’,DNA 链生成是单核苷酸 5’-磷酸基团向核酸链的 3’-OH 发起进攻。
- 磷酸 phosphate
1.2. DNA 表示方法
- 脱氧核苷酸的排列顺序称为 DNA 序列,又叫 DNA 的一级结构(如 TCA, 默认方向是 5’→3’)
- DNA 分子大小表示:bp, 碱基 (base 或 kilobase) 数目。
- 生物细胞内 DNA 总量:C 值,通常是指一种生物单倍体基因组 DNA 的总量,以每细胞内的皮克 (pg) 数表示。
1.3. 真核生物染色体 DNA 序列的特点
- 有重复序列
| / | 碱基组成 | 重复次数 | 占 DNA 总量 (%) | 基因类型 |
|---|---|---|---|---|
| 不重复序列 | 750-2000 | 1-几个 | 10-80% | 结构基因 |
| 中度重复序列 | 101-104 | 10-40% | rRNA, tRNA, 组蛋白基因 | |
| 高度重复序列 | 6-100 | 数百万次 | 10-60% | 卫星 DNA |
- 卫星 DNA (satellite DNA) : 又称随体 DNA。真核细胞 DNA 的一部分是不被转录的异染色质成分,其组成与主体 DNA 不同,因而可用密度梯度沉降平衡技术如氯化铯梯度离心将它与主体 DNA 分离。通常是高度串联重复的 DNA。
- 编码遗传信息:DNA 序列不同,携带的遗传信息不同。
- 有些序列编码蛋白质或者 RNA,称为功能 DNA,不编码的称为非功能 DNA(现在发现,非编码区也会编码大量 RNA)。不同生物中功能 DNA 占基因组的比例不同。
- 基因是 DNA 上的一段编码某种多肽链或 RNA 的顺序,还包括不被转录和不被翻译的具有调节功能的顺序。编码多肽链和 RNA 的基因称为结构基因。
1.4. 基因组
是指一套染色体中的完整的 DNA 序列。
- 基因组可以特指整套核 DNA, 也可以用于包含自己 DNA 序列的细胞器基因组,如粒线体基因组或叶绿体基因组。线粒体 DNA 和细菌 DNA 类似,能引起人体免疫反应。
- 基因组的大小:单倍体染色体中所有 DNA 的长度。
- 基因数:基因组中包含的基因数。
- 基因的密度:每 Mb 基因组 DNA 中所包含的平均基因的数量。
- 基因组大小与生物的复杂性:随着物种复杂度增加,各类生物的基因总数增加,但是基因的密度 (density) 降低。
C 值反常现象 (C-value paradox)
指 C 值(基因组大小)往往与种系的进化复杂性不一致的现象,即基因组的大小与遗传复杂性之间没有必然的联系(目前原因不明),某些较低等的生物 C 值却很大,如一些两栖类物种的 C 值甚至比哺乳动物还大。
真核基因组的结构特征
- 真核生物基因组很大,通常比原核生物大得多。
- 真核基因组具有大量重复序列。
- 真核基因组的大部分为非编码序列 (>90%),是真核生物与细菌和病毒之间最主要的区别。
- 真核基因组的转录产物为单顺反子(编码一个基因片段)。原核生物是多顺反子(一个 mRNA 分子编码多个多肽链)。
- 真核基因是具有内含子结构的片段化基因。
- 真核基因组具有大量的顺式作用元件(启动子、增强子、消音器)。
- 真核基因组中存在大量的 DNA 多态性:单核苷酸多态性和串连重复序列多态性。
- 真核基因组具有端粒 (telomere) 结构。保护线性 DNA 的完整复制、保护染色体末端和决定细胞的寿命等功能。
人类基因组
- 蛋白质编码区域只占人类基因组非常小的部分(~2-3%):30 亿对碱基,2-3 万个蛋白质基因。基因数目是果蝇的 2 倍,啤酒酵母的 4 倍。
- 其它 98% 的基因:人类基因组绝大部分都被转录成 RNA(非编码 RNA, ncRNA), 细胞内 ncRNA 的数量是编码 RNA 的上百倍。
- ncRNA 中约有 24% 为插入编码序列的内含子序列:人类基因平均每个基因有 7 个内含子。
- 生物的复杂性不由编码蛋白质的数目决定,而是被隐藏在它们所输出的非编码 RNA 内。
- ncRNA 是人与其他生物的主要差别:
人和黑猩猩的基因差别为 1‰, 来源于非编码 RNA 。
人和鼠的蛋白质编码基因 99%是共同的。
人个体间单倍体基因组的碱基差异 300 万个,其中 1 万个 (0.3%) 出现在蛋白质编码基因中,绝大多数存在于非编码 RNA。
ncRNA 功能
- 翻译:rRNAs and tRNAs
- mRNA 成熟:snRNA 识别剪接位点
- RNA 修饰:snoRNA 将尿苷(uridine)修饰变成假尿苷 (pseudo-uridine)
- 调节基因的表达与翻译:miRNAs
- DNA 复制:端粒酶 RNA 作为合成端粒重复序列的模板
1.5. DNA 在真核细胞中的结构
DNA 双螺旋结构 DNA double helix
- DNA 是由两条互相平行的脱氧核苷酸长链盘绕而成的。
- DNA 分子中的脱氧核糖和磷酸交替连接,排在外侧,构成基本骨架,碱基排列在内侧。
- 两条链上的碱基通过氢键连接形成碱基对。
碱基互补配对原则
- 在 DNA 分子中,嘌呤永远与密啶配对
- 腺嘌呤(A)= 胸腺喀啶(T) 2 个氢键
- 鸟嘌呤(G)≡ 胞啶(C)3 个氢键
DNA 构象
- 真核生物 DNA 以双螺旋的形式存在 A、B、Z 三种结构,B-DNA 是最常见的 DNA 构象
- 右手螺旋 DNAB-DNA: A-T 丰富的 DNA 片段A-DNA(对基因表达有重要意义) DNA-RNA(处于转录状态的 DNA)RNA-RNA (dsRNA)
- 左手螺旋 DNAZ-DNA 结构:1977 年 Rich 等人用人工合成的 dCGCGCG 六核酸进行 X-射线晶体衍射分析,观察到一种特殊的左手双螺旋结构,骨架走向呈锯齿状 (Zig-zag),称 Z-DNA。
A、B、Z-型 DNA 三种结构比较
| / | A | B | Z |
|---|---|---|---|
| 外型 | 粗短 | 适中 | 细长 |
| 螺旋方向 | 右手 | 右手 | 左手 |
| 螺旋直径 | 2.55nm | 2.37nm | 1.84nm |
| 碱基直升 | 0.23nm | 0.34nm | 0.38nm |
| 每圈碱基数 | 11 | 10 | 12 |
| 碱基倾角 | 20° | 0° | 7° |
| 大沟 | 很窄很深 | 很宽较深 | 平坦 |
| 小沟 | 很宽、浅 | 窄、深 | 较窄很深 |
特殊的 DNA 碱基序列可以有特殊的二级结构
- DNA 回文序列会形成 DNA 发夹结构 或 DNA 十字架结构
DNA 的三螺旋结构(H-DNA)
- Hoogsteen 碱基配对
- 三链结构:在 Waston-Crick 双碱基配对基础上,加入第三条链配对,形成 C≡G·C+(两个氢键)或 T=A·T 的三螺旋结构。
- DNA 三股螺旋结构(H 型-DNA)常出现在 DNA 复制、转录、重组的起始位点或调节位点,如启动子区。
- 第三股链的存在可能使一些调控蛋白或 RNA 聚合酶等难以与该区段结合,从而阻遏有关遗传信息的表达。
DNA 的四螺旋结构
- 真核生物 DNA 3’-末端(端粒)是富含 GT 的多次重复序列,因而自身形成了折叠的四链结构。
DNA 超螺旋结构(superhelix 或 supercoil)
- 正超螺旋:卷取方向与双螺旋相同
- 负超螺旋:卷取方向与双螺旋相反
- 意义: 使 DNA 形成高度致密状态从而得以装入核中;推动 DNA 结构的转化以满足功能上的需要。如负超螺旋分子所受张力会引起互补链分开导致局部变性,利于复制和转录。
- 研究细菌质粒 DNA 时发现,天然状态下该 DNA 以负超螺旋为主,稍被破坏即出现开环结构,两条链均断开则呈线性结构。在电场作用下,相同分子质量的 超螺旋 DNA 迁移率 > 线性 DNA 迁移率 > 开环的 DNA 迁移率,以此可判断质粒结构是否被破坏。
苏木精-伊红染色法(HE 染色) hematoxylin-eosin staining
- 石蜡切片技术中常用的染色方法之一。
- 苏木精染色液呈碱性,主要使细胞核内的染色质和细胞质内的核酸呈紫蓝色;
- 曙红是一种酸性染料,主要将细胞质和细胞外基质中的成分染成红色。
1.6. 染色体 chromosome
- 真核生物 DNA 的存在形式:在细胞周期的大部分时间里,DNA 以松散的染色质 (chromatin) 形式存在,在细胞分裂期,则形成高度致密的染色体 (chromosome)。
- 原核与真核细胞染色质的比较原核细胞中:DNA 存在于称为拟核 (nucleoid) 的结构区。每个原核细胞一般只有一个染色体,每个染色体含一个双链环状 DNA。真核细胞中:DNA 主要集中在细胞核内,线粒体和叶绿体中均有各自的 DNA。
- 真核染色体的特点:分子结构相对稳定;能够自我复制以保持父母和孩子之间的连续性;能够指导蛋白质合成,从而控制整个生命过程;能够产生可遗传的变异。
- 真核细胞染色体的组成DNARNA:尚未完成转录而仍与模板 DNA 相连接的;ncRNA 参与中心粒的形成等功能。组蛋白:8 聚体结构。非组蛋白
Histones
- 与 DNA 组成染色质(chromatin)的最小单位核小体 (nucleosome)。
- 有 H1、H2A、H2B、H3 及 H4 五种,是染色体的结构蛋白。
- H1 最不保守,位于染色质接头处;其它组蛋白进化上极端保守,位于染色质核心区。
- 无组织特异性。
- 肽链上氨基酸分布的不对称性:碱性氨基酸分布在 N 端;疏水基团在 C 端。
- H3 、H4 存在较普遍的修饰作用:甲基化、乙酰化、磷酸化、泛素化等。组蛋白 N 端尾巴的修饰位点主要在 K(lys 赖氨酸)和 R(Arg 精氨酸)(碱性氨基酸,带正电) 上。S(丝氨酸 Ser,带羟基)会发生磷酸化。
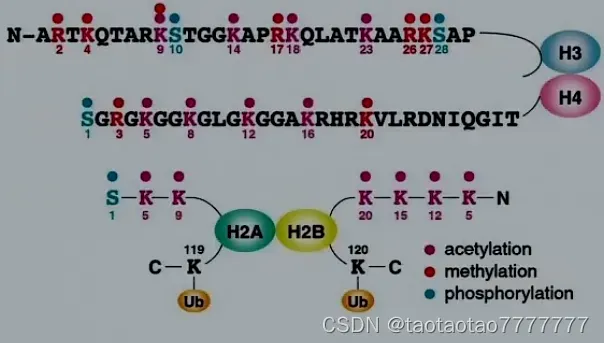
– 酸性氨基酸(负电):D(天冬氨酸 Asp)、E(谷氨酸 Glu)
– 20 种氨基酸缩写 [0] - 组蛋白的修饰改变染色质的功能**赖氨酸 K **的乙酰化(形成 50 种异构体);丝氨酸 S(H3, H4, H2B)的磷酸化;K 和 R(H3 和 H4)的甲基化修饰;其他修饰:如泛素化和 ADP-核糖基化。四种核心组蛋白尾部携带巨大的表观遗传信息量。
组蛋白甲基化
- 组蛋白的甲基化修饰是最稳定的,并且发生在组蛋白依赖性
关于氨基酸和精氨酸残基:
**赖氨酸 K **残基能够发生单、双、三甲基化;**精氨酸 R **残基能够单、双甲基化,这些不同程度的甲基化极大地增加了组蛋白修饰和调节基因表达的复杂性。 - 由组蛋白甲基化转移酶 (histonemethyl transferase, HMT)
Finish. - 组蛋白的甲基化与基因激活和基因沉默有关。
- 调节转录的位置,例如启动子,容易被修饰。
组蛋白乙酰基转移酶/组蛋白去乙酰化酶
Histone acetyltransferase (HAT) / Histone deacetylase complexes (HDAC)
- 乙酰化:加入乙酰基 CH3-CO-或 Ac-
- HAT/HDAC 催化 H3、H4 特定**赖氨酸 K **残基的乙酰化/去乙酰化修饰,实现乙酰化水平的动态平衡;
- 特定基因位点的组蛋白乙酰化和去乙酰化以非随机、位置特异性的方式发生;
- 乙酰化/去乙酰化修饰影响染色质结构和基因活化;
- 超乙酰化:转录激活;低乙酰化:转录抑制。
组蛋白乙酰化/去乙酰化参与的功能(与转录和结构相关)
- 转录激活、转录延伸
- DNA 修复、拼接、复制
- 染色体组装
- 基因沉默
- 某些疾病的形成
- 细胞信号转导
- 基因组的全局乙酰化
组蛋白的其他修饰
- 磷酸化、腺苷酸化、泛素化、ADP 核糖基化等不稳定的修饰方式。
- 各种修改之间也存在相互关系。
- 多种修饰的组合可以更灵活地调控染色质的结构和功能。这些可识别的修饰称为组蛋白代码。
- 组蛋白的泛素化 / 去泛素化:泛素化:加入泛素 (ubiquitin) 蛋白,调控组蛋白的活性,调控非组蛋白的降解。泛素蛋白连接酶 / 去泛素化酶 Histone ubiquitinase/ deubiquitinase 负责组蛋白 H2A 和 H2B 的单泛素化。
组蛋白修饰的一般作用机制
- 通过影响组蛋白与 DNA 双链的亲和性,改变组蛋白与 DNA 的相互作用,从而改变染色质的疏松或凝集状态;
- 它通过影响转录因子对结构基因启动子的亲和力来进行基因调控。
核心组蛋白尾部修饰的意义
- 调节 DNA 包装和染色质结构,进而调节基因的表达。
- 组蛋白修饰是表观遗传信息的载体,这些信息决定了基因表达模式如何在一代细胞和下一代细胞之间保持(部分保留)。
- 在肿瘤、免疫、心血管等疾病的发生和预防中具有深远的意义。
non-histone
- 非组蛋白约为组蛋白总量的 60% – 70%,可能有 20 – 100 种(常见的有 15 – 20 种)
- 主要包括酶类 (RNA 聚合酶)、与细胞分裂有关的蛋白。
- 它也可能是染色质的结构部分。
- HMG 蛋白 (High mobility group protein), DNA 结合蛋白,能与 DNA 结合但不牢固,也能与 H1 作用;可能与 DNA 的超螺旋结构有关。相对分子量较低,占非组蛋白的 20%,染色质的 8%; 可能与 DNA 的复制、转录、修复和重组有关。
1.7. 染色质和核小体
- 由 DNA 和组蛋白组成的染色质纤维细丝,是许多核小体连成的念珠状结构。
- **核小体 (Nucleosome) **是染色质的基本结构单位,由 200bp DNA 和组蛋白八聚体组成。
- 146bp DNA +组蛋白八聚体 (Histone octamer)→核小体核心 (Nucleosome core)+ H1→染色小体 (Chromatosome, 166bp) + linker DNA→核小体 (Nucleosome, ~200 bp of DNA)
染色质结构存在的实验证据:
- 染色质 DNA 的 Tm 值比自由 DNA 高,说明在染色质中 DNA 极可能与蛋白质分子相互作用。
- 在染色质状态下,由 DNA 聚合酶和 RNA 聚合酶催化的 DNA 复制和转录活性大大低于在自由 DNA 中的反应。
- DNA 酶 I (DNaseI) 对染色质 DNA 的消化远远慢于对纯 DNA 的作用。
- 用小球菌核酸酶处理染色质以后进行电泳,便可以得到一系列片段,这些被保留的** DNA 片段均为 200bp **基本单位的倍数。
染色质的高阶结构是如何形成的?
(1) 组蛋白 H1 与核小体之间的接头 DNA 结合,诱导更紧密的 DNA 包裹在核小体周围;(H1 的加入导致形成更紧凑的核小体 DNA)
(2) 核小体阵列可以形成更复杂的结构: 30nm 纤维 (zigzag model)。
- 30nm 染色质纤维是由“四聚核小体”为结构单元的左手双螺旋结构。
- 30nm 纤维具有 6 个核小体/转,组织成螺线管。
- 组蛋白** H1 和组蛋白 N 末端尾部**是形成 30nm 纤维所必需的。
- 这种纤维是间期染色质和有丝分裂染色体的重要组成部分。
(3) DNA 的进一步压缩形成大的核小体 DNA 环
原核基因组的特征
- 原核生物的基因组很小,大多只有一条染色体,且 DNA 含量少。如大肠杆菌 DNA 的相对分子质量仅为 4.6X106bp, 其完全伸展总长约为 1.3mm, 含 4000 多个基因。
- 原核生物基因主要是单拷贝基因,只有很少数基因(如 rRNA 基因)以多拷贝形式存在;
- 整个染色体 DNA 几乎全部由功能基因与调控序列所组成;
- 几乎每个基因序列都与其编码的蛋白质序列线性对应。
原核细胞 DNA 的特点
- 结构简炼:原核 DNA 分子的绝大部分是用来编码蛋白质的,只有很小一部分控制基因表达的序列不转录。这些不转录 DNA 序列通常是控制基因表达的序列。
- 存在转录单元:原核生物 DNA 序列中功能相关的 RNA 和蛋白质基因,往往从集在基因组的一个或几个特定部位,形成转录单元,并转录产生含多个 mRNA 的分子,称为多顺反子 mRNA。
- 有重叠基因:一些细菌和动物病毒存在重叠基因,同一段 DNA 能携带两种不同蛋白质的信息。
如何证明 DNA 和蛋白质分子之间发生了相互作用?
- 细胞外: EMSA 凝胶阻滞实验(电泳)足迹实验甲基化干扰试验ChIP-seq酵母单杂交技术核酸适配体双偏振干涉技术
- 细胞内:活细胞成像(荧光)
2. DNA 的复制 (DNA Replication)
2.1. DNA 复制的基本特点
- 半保留复制实验证据:密度梯度离心,当不同颗粒之间的沉降系数存在差异时,在一定离心力的作用下,颗粒各自以一定速度沉降,形成密度梯度不同区域的区域.
- 有复制起点和复制子复制起点:有特殊的复制起点,不同生物具有不同的复制起点数目和复制子数目复制子:一般把生物的复制单位称为复制子 (replicon) ,一个复制子只含有一个复制起点。细菌、病毒和线拉体的 DNA 分子都是作为单个复制子完成复制的。真核生物基因组包含多个复制子,可以同时在多个复制起点上进行复制。复制叉:DNA 分子的解链看起来像一个向一个方向生长的叉子 (fork)复制泡:被复制的区域看起来像气泡
- 双向等速复制大肠杆菌染色体复制实验 (1963,John Cairns)
将细菌培养在 3H 标记的培养基中,等细胞进入复制中期,温柔地裂解细胞,将细胞裂解液涂在电镜网格上将样品长时间( 2 个月)曝光到 X 胶片。
实验结果:大肠杆菌有环形染色质;大肠杆菌有单一的复制起始点;在大肠杆菌中复制和解链是同时发生的。双向复制(Bidirectional Replication)
无论是原核生物还是真核生物,DNA 的复制主要是从复制起点开始,以双向等速的复制方式进行。
复制叉以 DNA 分子上某一特定顺序为起点,向两个方向等速生长前进。 - 半不连续复制DNA 合成方向
DNA 双螺旋的两条链是反向平行的,因此两个模板的极性不同。
所有已知 DNA 聚合酶的合成方向是从 5’到 3’:DNA 的合成方向是从 5’到 3’。半不连续复制模型 (semi-discontinous replication) (等速复制) 用 3H 脱氧胸苷短时间标记后提取 DNA, 得到不少平均长度为 2-3kb DNA 片段。用 DNA 连接酶温度敏感突变株进行实验,在连接酶不起作用的温度下,有大量小片段累积,说明复制过程中至少有一条链首先合成较短的片段,然后再生成大分子 DNA。前导链的连续复制和滞后链的不连续复制,称为 DNA 的半不连续复制。短的半不连续片段被称为冈崎片段。细菌中大约 1000-2000 nt 长,真核生物中人约 200 nt 长。
2.2. DNA 聚合酶以及复制体系
- DNA 复制/合成体系:dNTPs: dATP, dTTP, dGTP, dCTP (脱氧三磷酸核苷)亲代 DNA templateDNA polymerase (Kornberg enzyme)Mg2+ (optimizes DNA polymerase activity)
- DNA 合成反应的主要特点DNA 聚合酶 Ⅰ 催化脱氧核糖的 3’-OH(在最后一个核首酸上)和 dNTP 的 5’-磷酸之间形成磷酸二酯键。该反应的能量来自 dNTP 的三个磷酸基团中两个磷酸的释放。DNA 聚合酶在延长过程的每个步骤“找到”正确的互补 dNTP。速率 ≤800 dNTPs/秒;错误率低合成方向是 5’到 3’
2.3. DNA 复制的基本过程
复制的起始 (initiation):在复制的起始原点处形成复制叉,复制叉能移动。
- 复制时,双链 DNA 在酶的作用下解链为两股单链分别进行,复制起点呈叉子形状,被称为复制叉 (Replication fork)
- 参与 DNA 复制起始和引发的蛋白质DNA 解旋酶 (DNA helicase):催化 DNA 双链的解链过程。单链 DNA 结合蛋白 (single strand DNA binding protein, SSBP): 以四聚体形式存在于复制叉处,只保持单链的存在,并不能起解链作用。DNA 拓扑异构酶 (DNA topoisomerase):消除 DNA 双链的超螺旋堆积。引物酶 (primase):是一种 RNA 聚合酶,能合成一小段 RNA 片段作为 DNA 复制的引物。引物是一个短序列(通常是 RNA),它与一条 DNA 链配对,并为 DNA 新链的合成提供 3’-OH 末端(作为 DNA polymerase Ⅲ的起始序列)。
- DNA 聚合酶的共同点: 都以脱氧三磷酸核苷 dNTP 为底物都需要 Mg2+ 激活聚合时都必须有模板链和具有 3’-OH 末端的引物链链的延伸方向为 5’到 3
- 复制的引发:DNA 聚合酶只能延长依存在的 DNA 链,不能从头合成 DNA 链,需要 3’-OH 末端起始复制。
DNA 链的延伸 (elongation)
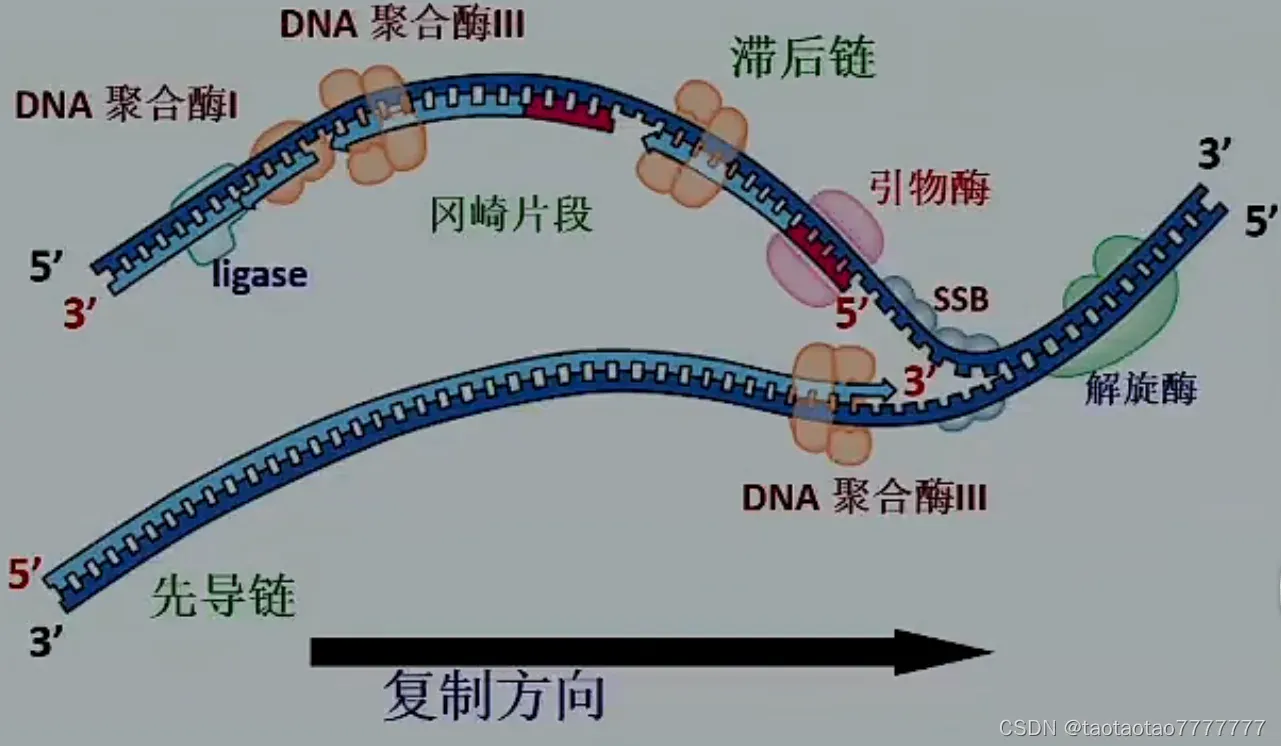
- DNA polymerase Ⅲ 的限制:只能加到已经存在的 DNA 或者 RNA 链的 3’端。
- 前导链:连续合成
- 滞后链:先合成冈崎片段,再通过**连接酶 ligase **连接起来。
- DNA polymerase Ⅰ具有 5’-3’外切酶活性,3’-5’合成活性。去除 RNA 引物并替换为 DNA 核苷酸(去除的末端由端粒酶合成)。合成速度 20bases/秒,主要作用是切除引物(移去不正常碱基)、编辑校对&改正错误碱基、修复错配碱基。
- DNA polymerase Ⅲ合成速度 1000bases/秒(速度快=lots of typos 错误多),主要作用是合成 DNA。
- DNA 链的延伸需要的蛋白质: DNA 聚合酶滑动夹 (Sliding DNA clamp):滑动夹环绕由相关 DNA 聚合酶产生的新复制的 DNA,能显著提高 DNA 的持续合成能力。RNA 酶 (RNase H 等 ):在复制完成后切除 RNA 引物。DNA 连接酶 (DNA ligase):通过生成 3’5’-磷酸二酯键连接两条 DNA 链。新合成 DNA 上 RNA 引物的去除: RNase H 移除几乎所有的 RNA 引物,只保留与 DNA 末端相连的核苷酸。5’核酸外切酶去除最后的核苷酸。DNA 聚合酶填补空隙,剩下一个 3’羟基和 5’磷酸基团之间的缺口。DNA 连接酶修复缺口。
复制的终止 (termination)
- Ter-Tus 终止复合物参与复制的终止:当复制叉迁移遇到 20bp 重复性终止子序列 (Ter) 时,Ter-Tus 终止复合物阻止复制叉的迁移。等到相反方向的复制叉到达后在 DNA 拓扑异构酶 V 的作用下复制叉解体,释放子链 DNA。
- 子代 DNA 分子的分离:Ⅱ型拓扑异构酶用来分离子代 DNA 分子;对于环状染色体,Ⅰ型拓扑异构酶催化其中一个子分子的断裂并允许第二个子分子通过断裂。
2.4. 原核生物 DNA 的复制
- 复制特点有多个复制起始点。复制叉的移动速度约 50bp/s, 人类 DNA 中每隔 30000-300000 就有一个复制起始位点。在全部完成复制之前,各个起始点上 DNA 的复制不能再开始。DNA 的复制只在 S 期进行。复制子相对较小,为 40-100k bp。
- 复制起点细菌染色体的复制是作为一个单位从唯一的复制起点开始,双向进行的。大肠杆菌 (E. Coli) 的 oriC 的复制原点:大肠杆菌基因组的复制原点位于天冬酰胺合酶和 ATP 合酶操纵子之间,全长 245 bp, 称为 oriC。
- 复制过程(由大肠杆菌 oriC 复制起始点处引发的 DNA 复制过程)大约 20 个 DnaA 蛋白在 ATP 的作用下与 oriC 处的 4 个 9 bp 保守序列相结合。在 Hu 蛋白和 ATP 的共同作用下,DnaA 复制起始复合物使 3×13 bp 直接重复序列变形,形成开链。DnaB (解链酶)六体分别与单链 DNA 相结合(需要 DnaC 的帮助)进一步解开 DNA 双链。与引物结合, 起始 DNA 复制。
- DNA 聚合酶大肠杆菌中主要有 DNA 聚合酶Ⅰ、Ⅱ、Ⅲ、Ⅳ 和 Ⅴ。DNA 聚合酶Ⅰ(coded by polA) 不是复制大肠杆菌染色体的主要聚合酶:有 3’→5’核酸外切酶活性,保证 DNA 复制的准确性。它的 5’→3’核酸外切酶活性可用来除去岗崎片段 5’端 RNA 引物,同时利用 5’→3’ DNA 合成活性去除岗崎片段缺口。还参与 DNA 损失的修复。DNA 聚合酶 Ⅱ(coded by polB) 活性很低,只有 DNA 聚合酶的 5%,也不是复制中的主要酶。生理功能主要是修复 DNA,具有 3’→ 5’核酸外切酶活性。DNA 聚合酶Ⅲ(coded by polC) 是大肠杆菌 DNA 复制中链延长反应的主导聚合酶:聚合活性较强,DNA 聚合酶Ⅰ的 15 倍,聚合酶Ⅱ的 300 倍。合成速度:5 万 nt/min。包含 7 种不同的亚单位和 9 个亚基,以二聚体的形式发挥生物活性。具有 3’→5’核酸外切酶活性。
- 复制的几种主要方式θ型:大肠杆菌质粒 DNA 双向复制模式滚环型 rolling circle:属于特定环状 DNA 分子的复制方式(单向复制),环状 DNA 可以通过滚环式复制产生多单元单链 DNA,是噬菌体 DNA 在细菌中最通常的一种复制方式; 滚环型复制过程:环状双链 DNA 的+链被内切酶切开(单缺口);以-链为模板,DNA 聚合酶以+链的 3’端作为引物合成新的+链,原来的+链 DNA 分子的 5’端与-链分离;+链的 3’端继续延长;引发酶以离开的+链为模板合成 RNA 引物,DNA 聚合酶以+链为模板合成新的-链;通常滚环复制的产物是一多聚物,其中大量单位基因组头尾相连。复制过程的特征:复制是单方向不对称的;产物是单链 DNA, 但可通过互补链的合成转变为双链;子代 DNA 分子可能是共价连接的连环分子;连环分子随后被切成与单个基因组相对应的片段。 D-环型 D-loop:单向复制的特殊方式 首先在动物线粒体中被发现。植物叶绿体基因组也是靠 D 环复制的。一条短 RNA 与一条 DNA 互补,取代了该区域原来互补的 DNA 链,使得两条链的合成高度不对称,一条链上迅速合成出互补链,另一条链则为游离的单环。两条链的复制都是从两个独立的起点先后起始的;复制的起始是由一条或两条(链)替代环促使的。
原核生物、真核生物和病毒复制起点的共同特征
- 起始位点是包括多个短重复序列的独特 DNA 片段;
- 多聚体 DNA 结合蛋白专一性识别这些短的重复序列;
- 起始位点旁侧序列是丰富的 AT,能使 DNA 螺旋容易解开。
2.5. 真核生物 DNA 的复制
- 真核生物 DNA 的复制子称为自主复制序列 (ARS, autonomously replicating sequence),长约 150bp,包括数个复制起始必需的保守区。
- 真核生物 DNA 复制的起始需要起始点识别复合物 (origin recognition complex, ORC) 参与,ORC 由 6 种蛋白质组成,结合 ARS。
- DNA 聚合酶真核生物 DNA 聚合酶有 15 种以上,在哺乳动物细胞中主要有 5 种 DNA 聚合酶,分别称为 DNA 聚合酶 α、β、γ、δ和ε。DNA 聚合酶 α 主要参与引物合成。DNA 聚合酶 β 活性水平稳定,主要在 DNA 损伤的修复中起作用。DNA 聚合酶 δ 主要负责 DNA 的复制。DNA 聚合酶 ε 与后随链合成有关,在 DNA 合成过程中核苷切除以及碱基的切除修复中起着重要的作用。DNA 聚合酶 γ 在线粒体 DNA 的复制中发挥作用。真核生物中还存在 ζ、η、τ和κ 等几种 DNA 聚合酶,它们承担着修复损伤的功能,但这些修复酶的忠实性都很低。δ 和 ε 具有 3’→5’外切酶活性。
真核生物 DNA 聚合酶的特性比较
| 性质 | α | β | γ | δ | ε |
|---|---|---|---|---|---|
| 亚基数 | 4 | 1 | 2 | 2-3 | ≥1 |
| 细胞内分布 | 核内 | 核内 | 线粒体 | 核内 | 核内(?) |
| 功能 | DNA 引物合成 | 损伤修复 | 线粒体 DNA 复制 | 主要 DNA 复制酶 | DNA 复制(?) |
| 3’→5’外切 | 无 | 无 | 有 | 有 | 有 |
| 5’→3’外切 | 无 | 无 | 无 | 无 | 无 |
3. 基因组 DNA 序列的变异与修复
3.1. DNA 复制过程中的变异与修复
- 复制的精确性 (Replication fidelity)对于遗传信息从上一代精确传到下一代很重要。在大肠杆菌中 DNA 复制的错配率很低,DNA 聚合酶的校对功能 (proofreading function) 保证了复制的精确性。校对是指用于纠正蛋白质或核酸合成中的错误的任何机制,其涉及在将各个单元添加到链中之后对其进行详细检查。DNA 聚合酶含有 DNA 聚合 (Polymerizing,E) 和校读 (proofreading, P)两个不同的位点。大肠杆菌 DNA 聚合酶的校对功能: 当在 DNA 合成期间掺入不正确的碱基时,聚合酶暂停,然后将生长链的 3’末端转移至核酸外切酶位点来去除错误碱基。然后 3’端翻转回聚合酶位点,正确复制该区域。大肠杆菌 DNA 聚合酶Ⅰ,Ⅱ和Ⅲ具有 3’→5’核酸外切酶活性,因此具有校对活性。在动物细胞中,δ和ε DNA 聚合酶,而不是聚合酶 α, 都具有校对活性。
- 染色体侵蚀:因为 DNA 聚合酶只能添加到现有 DNA 链的 3’端,所以在去除 RNA 引物后 DNA 聚合酶/连接酶不能填充染色体末端的缺口。如果这个差距没有填补,每轮复制后染色体会变短!solution:真核生物在其染色体末端有连续重复非编码序列(端粒)(保护帽,细胞分裂限制在 50 次以内)。端粒酶 (Telomerase) 与末端端粒重复序列结合并催化添加新的重复序列。通过延长染色体来补偿。端粒酶活性的缺失或突变导致染色体缩短和有限的细胞分裂。
端粒 Telomere
- 端粒 是人所有染色体末端的重复 DNA 序列
- 它们包含数千个六核苷酸序列 TTAGGG 的重复序列
- 在人类中有 46 条染色体,因此有 92 个端粒(每端一个)
- 人类中一条链上重复的富含 G 的序列:(TTAGGG)n
- 重复可以长达数千个碱基对。在人类中,端粒重复平均为 5-15kbp
- 端粒特异性蛋白质,例如 TRF1 和 TRF2, 结合重复序列并保护末端。没有这些蛋白质,端粒受到 DNA 修复途径的作用,导致染色体融合。
- 端粒的功能: 保护染色体。将一条染色体与另一条染色体分开。没有端粒,染色体的末端将被“修复”,导致染色体融合和大量的基因组不稳定性。端粒也被认为是调节单个细胞分裂次数的“时钟”。每次 DNA 复制时,端粒序列都会缩短。
端粒酶 Telomerase
- 端粒酶由与端粒重复序列互补的 RNA 和蛋白质组成
- 端粒酶合成端粒 DNA:端粒酶具有逆转录酶活性,其中的 RNA 与端粒配对,合成端粒 DNA。
- 人端粒酶的两个组成部分: 人 RNA 亚基 (hTR) 5-CUAACCCUAAC-3’人端粒酶逆转录酶 (hTERT)
- 功能: 专门的逆转录酶通过添加端粒来防止“缩短末端问题”仅复制其自身携带的一小部分 RNA需要 3’末端作为引物合成以 5’→3’方向进行合成一个重复然后重新定位自己活跃时提供细胞永生
- 端粒酶通过将端粒 DNA 添加到染色体末端起作用,从而补偿通常在细胞分裂时发生的端粒丢失。
- 身体中的大多数细胞都含有端粒酶,但它是“关闭的”,因此细胞在每次分裂时都会失去端粒并最终死亡。
- 在人类中,端粒酶在生殖细胞、血细胞、体外永生化细胞、绝大多数癌细胞和可能的一些干细胞中具有活性。
- 端粒酶与癌症:来自数百个独立实验室的实验证据表明,端粒酶活性几乎存在于所有人类肿瘤中,但不存在于肿瘤附近的组织中。因此,临床端粒酶研究目前的重点是开发准确的癌症诊断方法和新型抗端粒酶癌症治疗方法。
- 端粒酶和衰老:癌细胞不会老化,因为它们会产生端粒酶,使端粒保持完整。大量实验表明端粒与衰老之间存在直接关系,端粒酶具有延长寿命和细胞分裂的能力。
3.2. DNA 变异与修复
3.2.1 突变 mutation
突变:DNA 碱基序列的永久性、可遗传的改变。
突变原因
- 自发性的:未知原因;可能是一个致病因素,但尚未确定;在噬菌体和细菌中以相对恒定的速率进行:每代每个基因组 3-4×10-3。
- 诱发:物理或化学制剂、尼古丁、紫外线、药物、电离辐射等。
突变类型
- 核苷酸之间的突变Transition(转换): Purine 嘌呤 or pyrimidine 嘧啶 is replaced by the other. A↔G or T↔CTransversion(颠换): a purine is replaced by a pyrimidine or vice verse. 嘌呤被嘧啶取代,反之亦然。A↔T/C or G↔T/CSilent mutation 沉默突变:TGT→TGC Cys→Cys 氨基酸不发生改变Missense mutation 错义突变:TGT→TGG Cys→Trp 氨基酸发生改变Nonsense mutation 无义突变:TGT→TGA Cys→Stop 变成终止子
- 核苷酸的缺失或插入移码突变 frameshift:The elimination or addition of one or more nucleotides in a DNA region. It may cause frameshift (移码突变) producing a non-functional protein.
3.2.2 E.coli DNA 修复系统
- 错配修复 mismatch repair:修复错配。根据母链甲基化原则找出错配碱基,MutS-MutL 与碱基错配位点的 DNA 双链结合移动,发现甲基化 DNA 后由 MutH 切开非甲基化的子链。
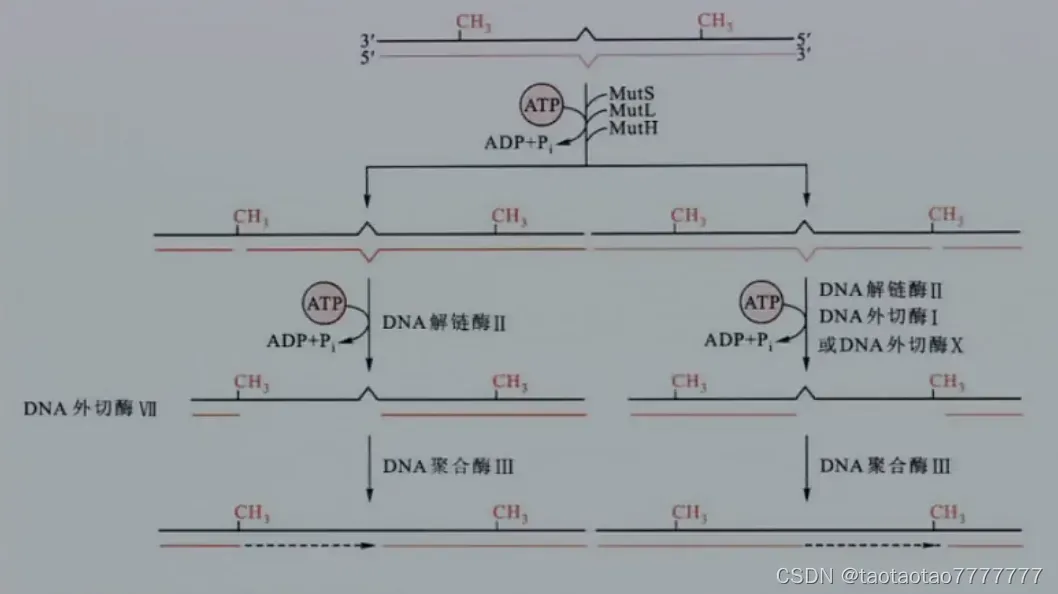 当错配碱基位于切口 3’下游端时,在 MutL-MutS、解链酶Ⅱ 、DNA 外切酶Ⅵ 或 RecJ 核酸酶的作用下,从错配碱基 3’下游端开始切除单链 DNA 直到原切口,并在 Pol Ⅲ和 SSB 的作用下合成新的子链片段;
当错配碱基位于切口 3’下游端时,在 MutL-MutS、解链酶Ⅱ 、DNA 外切酶Ⅵ 或 RecJ 核酸酶的作用下,从错配碱基 3’下游端开始切除单链 DNA 直到原切口,并在 Pol Ⅲ和 SSB 的作用下合成新的子链片段;
若错配碱基位于切口 5’上游端,则在 DNA 外切酶Ⅰ或 Ⅹ 的作用下,从错配碱基 5’上游端开始切除单链 DNA 直到原切口,再合成新的子链片段。 - 切除修复(碱基、核苷酸切除修复):Base-excision repair / nucleotide-excision repair 切除突变的碱基和核苷酸片段。
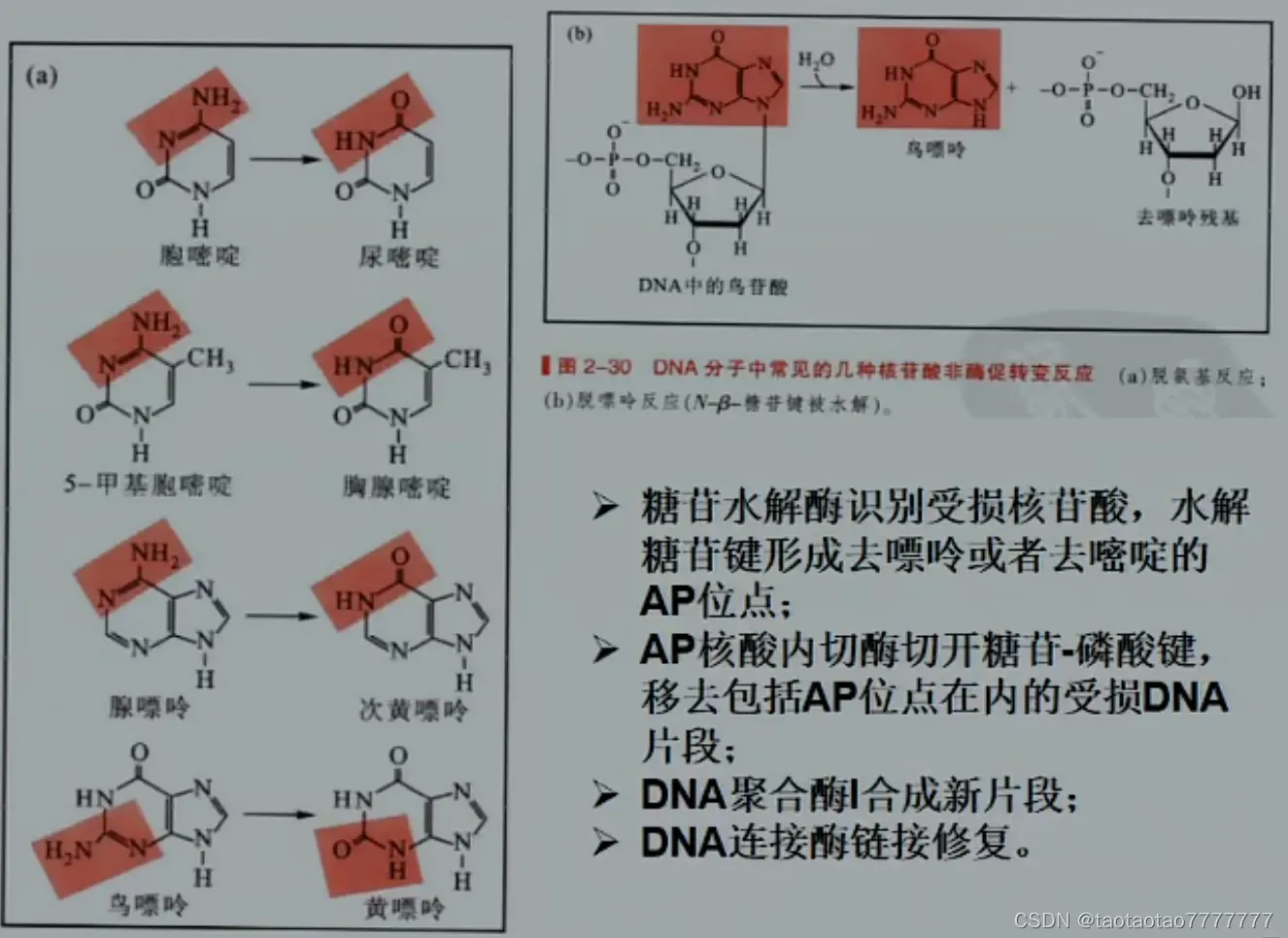 DNA 分子中常见的核苷酸非酶促转变反应:(a) 脱氨基反应(嘌呤上的氨基被氧化);(b) 脱嘌呤反应 (N-β-糖苷键被水解)。
DNA 分子中常见的核苷酸非酶促转变反应:(a) 脱氨基反应(嘌呤上的氨基被氧化);(b) 脱嘌呤反应 (N-β-糖苷键被水解)。 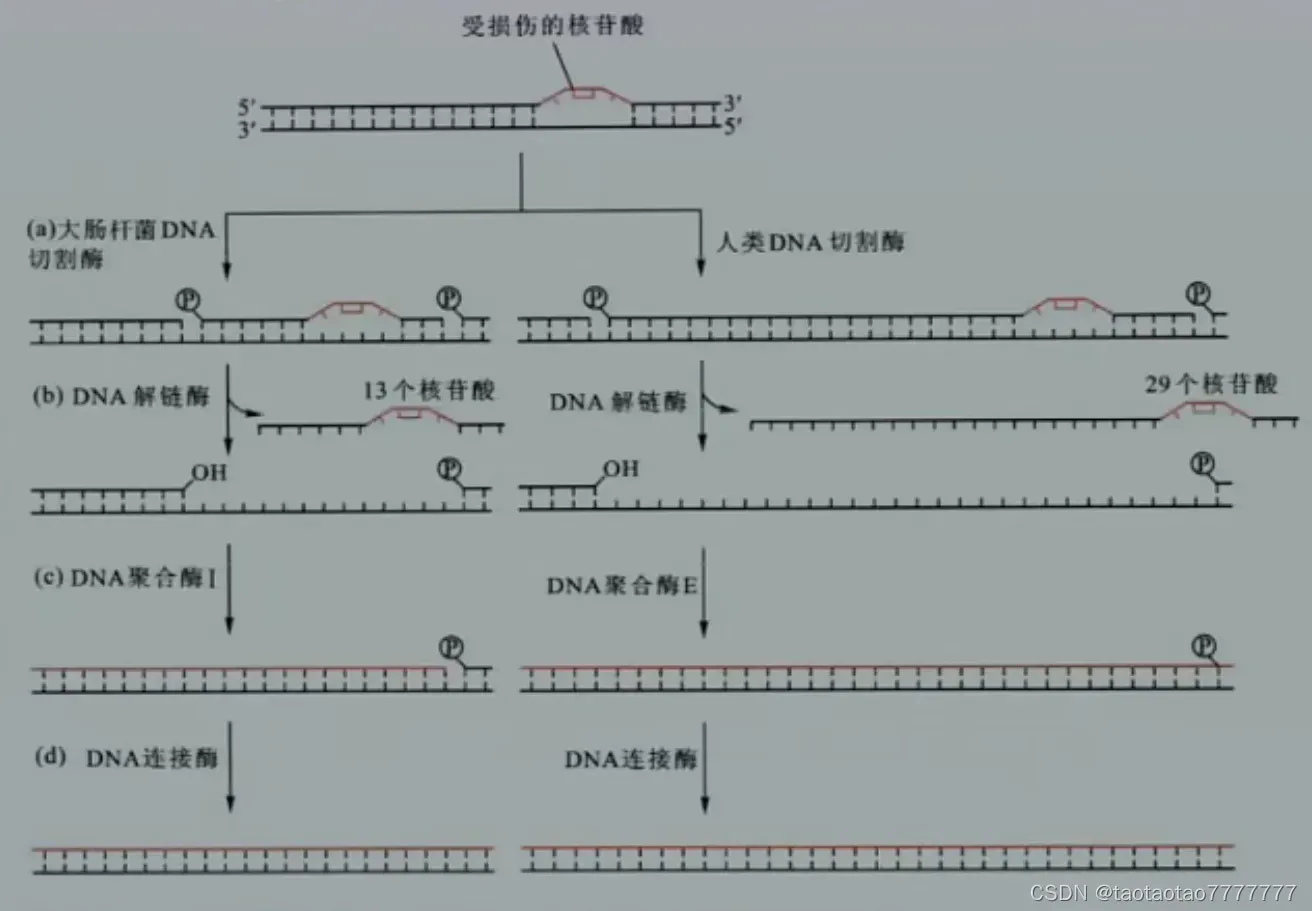 大肠杆菌(左)和人类细胞(右)中的核苷酸切除修复过程示意图
大肠杆菌(左)和人类细胞(右)中的核苷酸切除修复过程示意图
在原核生物中受损伤核苷酸3’端的第** 5 位,5’端的第 8 位磷酸糖苷键分别被 DNA 切割酶切开。
在人类细胞中,受损伤核苷酸3’端第 6 位,5’端的第 22 **位磷酸糖苷键分别被 DNA 切割酶切开。 - 重组修复 recombinant repair:复制后的修复,重新启动停滞的复制叉。
- DNA 直接修复 direct repair:修复嘧啶二体或甲基化 DNA。
- SOS 系统: 细胞 DNA 受到损伤或复制系统受到抑制时,细胞为求生存而“生的一种应急措施。诱导 DNA 损伤修复、诱变效应、细胞分裂的抑制以及溶原性细菌释放噬菌体等,导致变异。
3.2.3 SNP
(1)SNP 定义
- 单核苷酸多态性
(SNP, Single Nucleotide Polymorphism)
指基因组 DNA 序列中由于单个核苷酸(A,T,C 和 G)的突变而引起的多态性。 - SNP 是基因组中最简单最常见的多态性形式,具有很高的遗传稳定性。
- SNP 检测和分析技术成为第三代遗传标记。
- 一个 SNP 表示在基因组某个位点上一个核苷酸的变化: 转换(C⬅➡T,在其互补链上则为 G⬅➡A) 占 SNP 总量的** 2/3**。嘧啶⬅➡嘧啶,嘌呤⬅➡嘌呤颠换 (C⬅➡A/G,T⬅➡A/G) 嘧啶⬅➡嘌呤
- SNP 广泛存在于人类基因组中,其发生频率约为 1% 或更高。一个人类个体大约携带** 300 万-1000 万**个 SNPs。
(2)SNP 分类
根据 SNP 在基因组中的分布位置可分为三类:
1. 基因编码区 SNP (cSNP),20%
(1) 同义 cSNP (synonymous cSNP) 即 SNP 所导致的编码序列改变并不影响其所翻译的蛋白质的氨基酸序列,突变碱基与未突变碱基的含义相同;
(2) 非同义 cSNP (non-synonymous cSNP) 指碱基序列的改变可使以其为蓝本翻译的蛋白质序列发生改变,从而影响了蛋白质的功能。是导致生物性状改变的直接原因。
2. 基因调控区 SNP (pSNP) 影响基因表达量的多少
3. 基因间随机非编码区 SNP (rSNP)
(3)SNP 检测
SNP 检测技术最常见的是通过 DNA 测序法获得新的 SNP。
基因分型(genotyping) 是指利用数据库中已有的 SNP 进行特定人群的序列和发生频率的研究:
· 基因芯片技术
. Taqman 技术
· 分子信标(molecular beacons)技术
· 焦磷酸测序法( pyrosequencing)
(4)SNP 的应用
- 绘制人类单倍型图
已有超过** 150 万**个 SNP 被精确定位于各染色体上。将为我们精确定位复杂疾病,如糖尿病,癌症,心脏病,中风,哮喘等易感基因提供重要信息。 - SNP 与疾病易感基因的相关性分析
SNP 作为新一代遗传标记在人类疾病研究中显示出极高的潜在价值。已经发现了高血压、哮喘、类风湿关节炎、肺癌、前列腺癌等许多易感基因。 - 药物和药物设计指南
通过研究 SNP 与个体对药物敏感或耐受的相关性研究,可能阐明遗传因素对药效的影响,因此可能建立与基因型相关的治疗方案,对病人施行个性化用药。另外,随着 SNP 的研究与药物基因组学的结合,根据特定的基因型来设计药物将成为可能。
3.3. DNA 的转座 ★★★
3.3.1 转座的定义
- DNA 的转座,或称移位 (transposition),是由 可移位因子(transposable element)介导的 遗传物质重排 现象。
- 转座子(transposon, Tn) 是存在于染色体 DNA 上可自主复制和位移的基本单位。
3.3.2 转座模式
- 根据转座子 内部是否有标记基因 一般分为 插入序列(IS) 和复合型转座子两类;插入序列(IS) 在转座子的两侧有两个反向重复序列(倒置重复区),在靶 DNA 插入位点有两个短的正向重复序列(靶序列的顺式重复)。
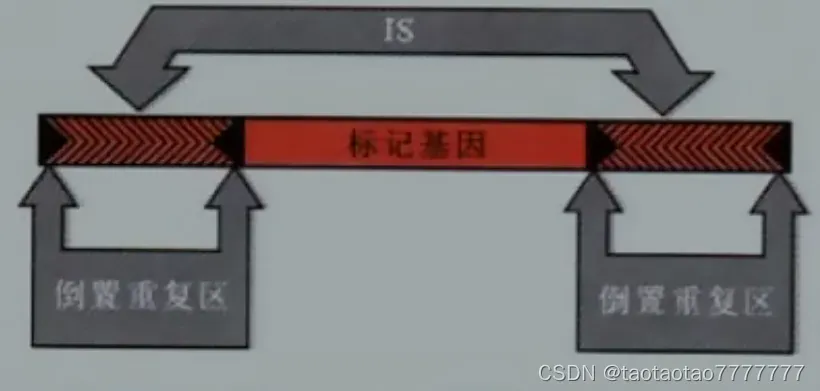
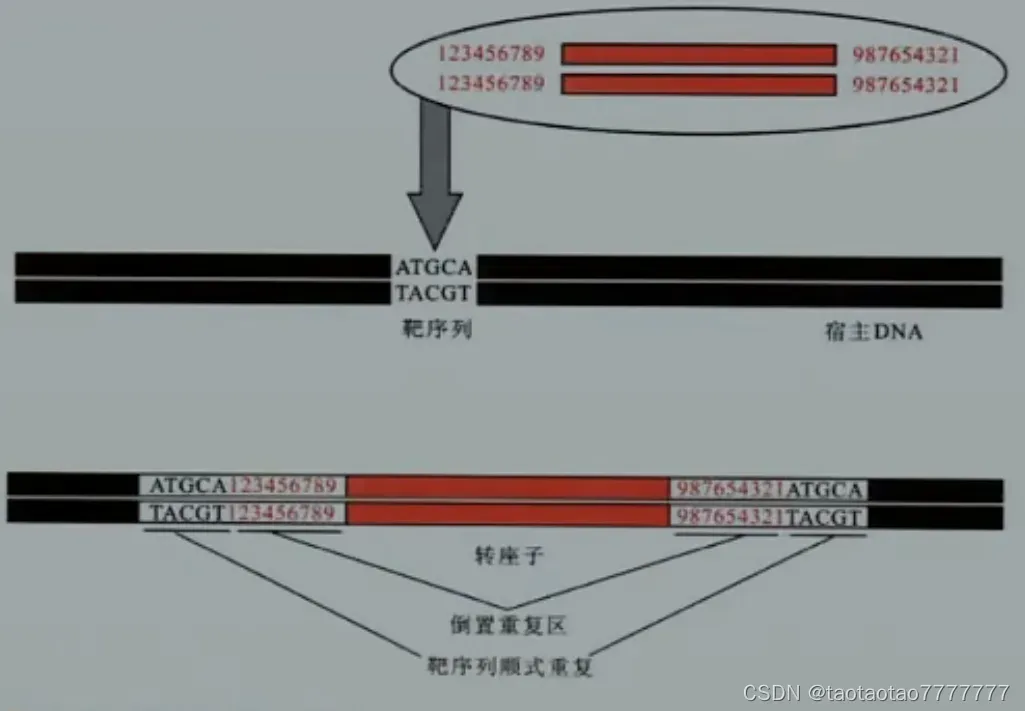 复合型转座子 的两侧各有一个** IS 序列**(IS 不能单独移动,带抗药性基因或者宿主基因一起转座),每个 IS 序列两侧各有倒置重复序列。
复合型转座子 的两侧各有一个** IS 序列**(IS 不能单独移动,带抗药性基因或者宿主基因一起转座),每个 IS 序列两侧各有倒置重复序列。
 TnA 类转座子:体积庞大 (>5kb)
TnA 类转座子:体积庞大 (>5kb)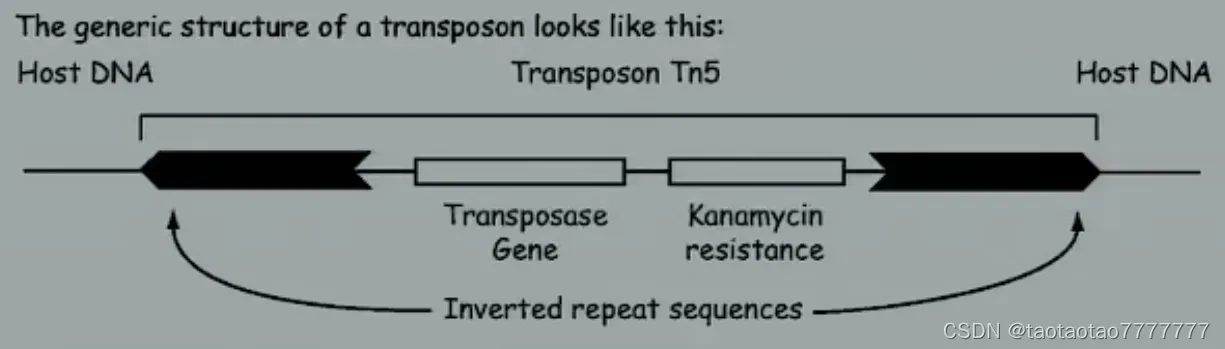 转座子组成功能转座酶 Transposase随机剪切目标 DNA 并将转座子末端拼接到目标序列上。转座的其他步骤由宿主酶完成。倒置重复序列 Inverted Repeats, IR这些序列直接作用于转座子的末端。注意,由于序列是倒置的,两端序列相同。筛选标记 Selectable Markers转座子被认为是通过向宿主细胞提供选择性优势而进化的。许多转座子携带给宿主带来抗生素耐药性 (antibiotic resistance) 或其他益处的基因。
转座子组成功能转座酶 Transposase随机剪切目标 DNA 并将转座子末端拼接到目标序列上。转座的其他步骤由宿主酶完成。倒置重复序列 Inverted Repeats, IR这些序列直接作用于转座子的末端。注意,由于序列是倒置的,两端序列相同。筛选标记 Selectable Markers转座子被认为是通过向宿主细胞提供选择性优势而进化的。许多转座子携带给宿主带来抗生素耐药性 (antibiotic resistance) 或其他益处的基因。 - 根据转座子是否移动,分为复制型(转座后保留原序列)和非复制型(转座后不保留原序列)两类。
3.3.3 真核转座子
- 转座子存在于所有生物体内,人类基因组中有 35% 以上的序列为转座子序列,其中大部分与疾病相关。一般** IS 转座频率** 10-3~10-4 /世代。
- 玉米中转座子 分为 2 类: 自主性转座子 (Ac) 和 非自主性转座子 (Ds)。自主性转座子可独立转座。非自主性转座子在有同家族的自主性转座子时可以发生转座,同家族的自主性转座子起着提供转座酶的作用。玉米转座子具有典型的 lS 特征——在转座子的两侧有两个反向重复序列,在靶 DNA 插入位点有两个短的正向重复序列。Ac 转座形成不稳定的基因突变,但不使染色体断裂;Ds在** Ac 帮助下转座,能使基因失活或者改变表达量,同时使染色体断裂**,引起缺失或者重组。
玉米中 Ac-Ds 系统(激活-解离系统)
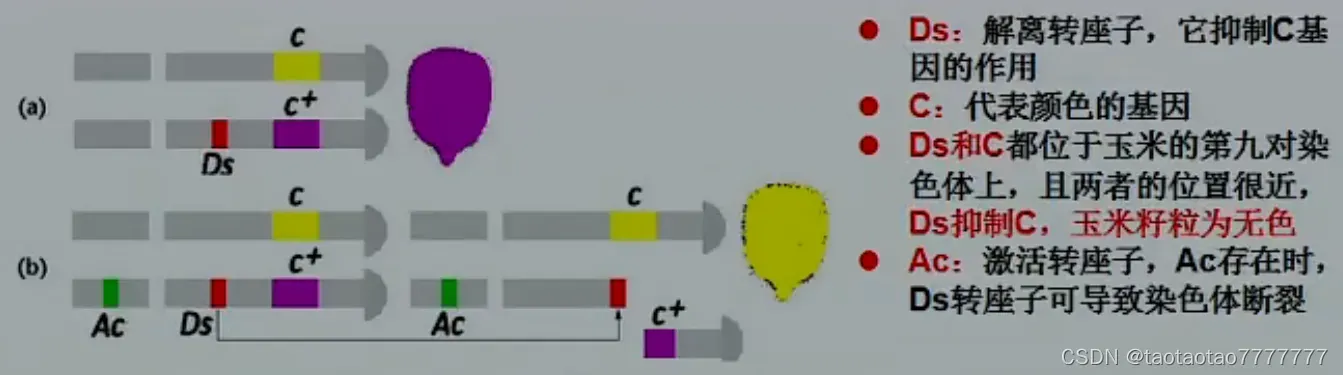
- 断裂的遗传标记是在 c 基因的表达:隐性 c 等位基因在个体种子(籽粒)中产生突变体无色表型,并且显性 c+等位基因产生野生型黑暗(紫色)表型。c/c=白色;c+/c=紫色
- 核心颜色等位基因/特征是“不稳定的”:C 与 Ds 基因之间容易发生断裂,使Ds 基因转座到原来染色体的其它部位或别的染色体,于是 C 恢复活性表现紫色。
- 如果在细胞中发生 c/c 向 c+/c 的转化,细胞将产生紫色色素和斑点。在发育的越早期发生转化,黑色部分越多。
- McClintock 得出“c”等位基因结果来自插入“C”基因的一种称为“Ds”的非自主转座子 (Ds =解离) 。
玉米中 Ac 自主和 Ds 非自主转座子的结构

- Ac 元件是自主的,Ds 是非自主转座子。
- Ac 为 4,563bp。具有 11bp ITR 和 1 个编码 807 个氨基酸转座酶的转录单位。
- Ac 激活 Ds; Ds 的长度和顺序不同,但具有与 Ac 相同的 ITR。
- 许多 Ds 元件是截短或重排的 Ac 版本,来自 Ac。
- Ac / Ds 受发育调节;Ac / Ds 转座仅在染色体复制期间。
3.4. DNA 突变和转座的应用
3.4.1 突变的应用
- 线虫的遗传筛查
线虫 (C. elegans) 是经典的遗传学模式动物:快捷的 DNA 显微注射;海量基因突变品系库;方便的 RNAi 操作;易于进行高通量遗传学筛选。
正向筛选 (forward, genome-wide mutagenesis):化学诱变筛选,基于遗传性状、荧光标记和蛋白定位的筛选。
1974 年 sydney Brenner 的论文 The Genetics of Caenorhabditis elegans 中,通过 EMS 诱变一共获得 300 多个线虫突变体,其中多数为隐性突变。突变表型涉及行为、运动和形态结构等方面,这些突变材料使得 100 多个基因得以表征,并被定位于 6 个连锁群。
反向筛选 (reverse, target-selected and gene-specific mutagenesis)
3.4.2 转座的应用
转座的遗传效应
① 转座引起插入突变,导致结构基因失活。
② 转座产生新基因。
③ 转座产生染色体畸变。
④ 转座引起生物进化。
使用转座子的非定向插入诱变
- 转座子是在转座酶的帮助下进出基因组的 DNA 序列,转座酶通常由转座子本身编码。
- 在 Mos1 转座酶存在下,外源转座子 Mos1 在 N2 种系中具有活性。
- 当激活时,内源转座子和 Mos1 随机插入基因组。
- 可以使用反向 PCR 或全基因组测序来鉴定被破坏的基因(通过转座子序列,找到临近的基因,即被影响的基因)。
3.4.3 靶向基因编辑
有缺口的基因组** DNA 修复后可能产生阅读框移位**。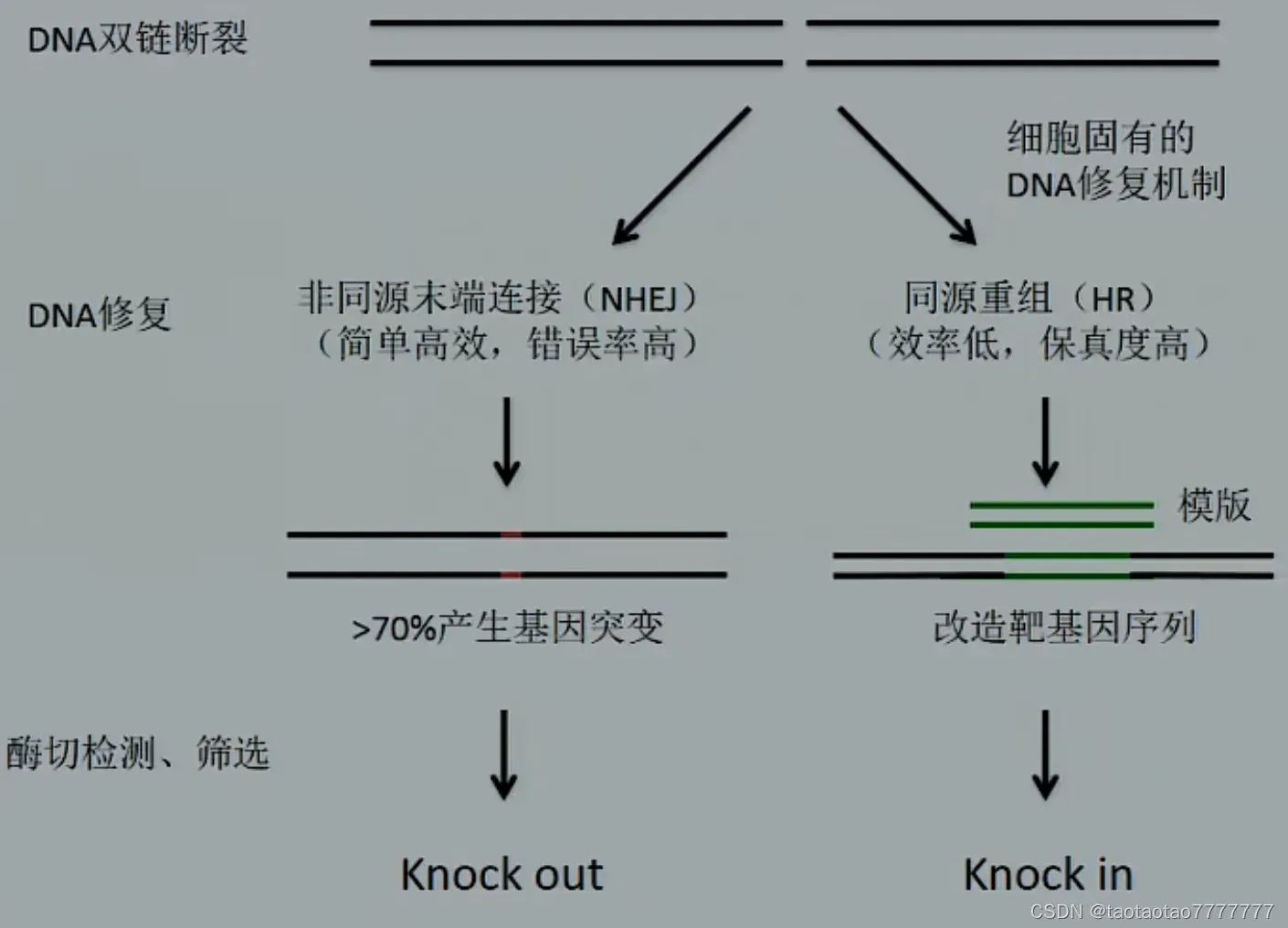
基于转座子的靶向编辑一 Mos1 excision-induced Transgene-Instructed gene Conversion (MosTIC)
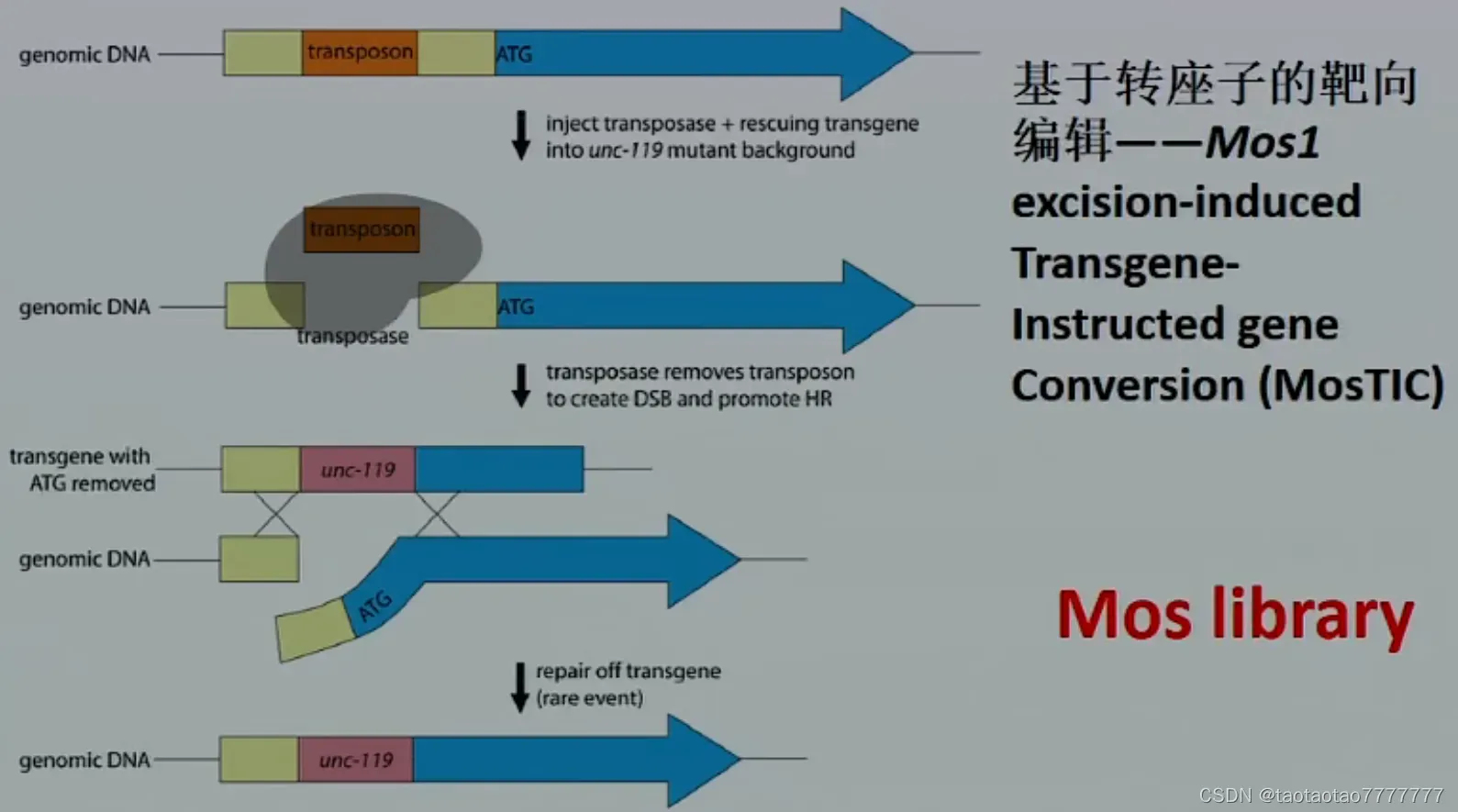
为了产生靶向基因缺失,将转座酶和含有选择标记 (unc-199) 的拯救转基因注射到 unc-119 突变体动物中,该动物在目的基因附近或目的基因中携带转座子。
通常,在目的基因附近携带单个 Mos1 转座酶插入的菌株来自 NemaGENETAG Consortium (Bazopo et al., 2012)。
双链断裂 (DSB) 后的同源定问修复以较低的概率发生。
在** MosDEL 的情况下,基因的一些部分被删除**,通常是基因的 5’区域。
在** MosTIC **中,转基因含有小的插入、缺失或点突变,其被整合到基因组 DNA 中。图中 unc-119(+) 被插入到基因组中。
基于酶的靶向编辑
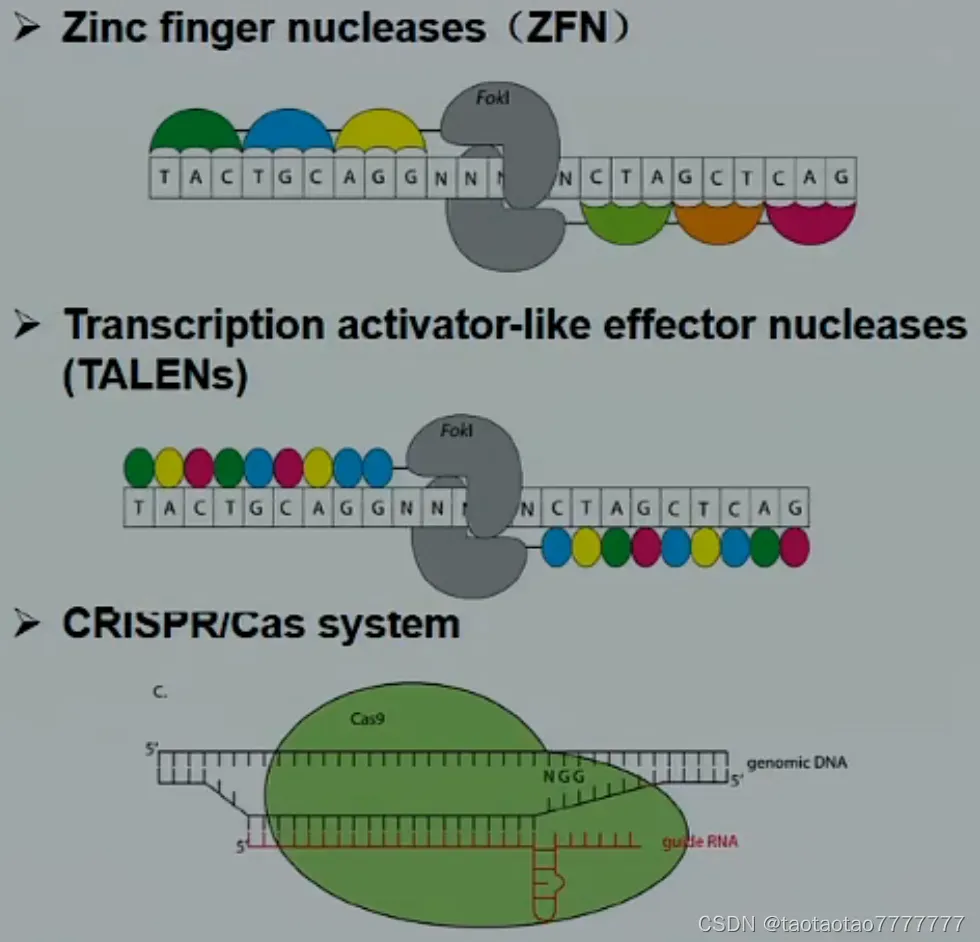
关键:具体识别
文章出处登录后可见!
