原文标题 :Decision Tree Hyperparameter Tuning in R using mlr
使用 mlr 在 R 中进行决策树超参数调整
了解如何使用 mlr 在 R 中执行超参数网格搜索

许多人通过学习和应用决策树算法进入数据科学之旅。这并不奇怪,因为该算法可能是最容易解释的算法,并且可以很好地模仿人类决策。
理解决策树还有另一个巨大的优势:它们是最著名的提升(Extreme Gradient Boosting)和装袋(随机森林)算法的基础,这些算法在 Kaggle 比赛中一举成名,解决了全世界无数的商业问题。
在您掌握了决策树是如何构建的以及它如何选择用于在数据中执行关键拆分的特征的细节之后,您会立即明白,当我们开始拟合它来解决问题时,需要做很多决策问题——即:[0]
- 树应该走多深?
- 拆分节点时应考虑的相关示例数量是多少?
- 我们会考虑多少个例子来做出决定?
- 考虑新拆分的最小基尼/熵增益阈值是多少?
所有这些问题似乎都有点武断。但是,您可能会注意到它们都与决策树的一个关键特性相关联——超参数:模型未学习但由用户参数化的一组值。
调整这些超参数对于实现所有机器学习算法的最终目标(泛化能力)至关重要。而且,在决策树中,可以说它们甚至更重要,因为基于树的算法对超参数空间中的微小变化非常敏感。
训练超参数是世界各地数据科学家和机器学习工程师的一项基本任务。而且,了解这些参数中的每一个的个人影响将使您信任并更好地解释您的表现。
在这篇文章中,我们将使用 R 和 mlr 库来优化决策树超参数。我还想向您展示如何可视化和评估每个参数对我们算法性能的影响。对于我们的示例,我们将使用 Kaggle 中提供的神话般的泰坦尼克号数据集。[0]
Let’s start!
Loading the Data
在加载数据之前,让我们调用代码的所有依赖项:
library(dplyr)
library(rpart)
library(rpart.plot)
library(Metrics)
library(mlr)
library(ggplot2)
library(plotly)Describing our dependencies:
- dplyr 执行一些数据整理任务。
- rpart 以适应决策树而无需调整。
- rpart.plot 绘制我们的决策树。
- 评估我们模型性能的指标;
- mlr 来训练我们模型的超参数。
- ggplot2 用于我们将做的一般情节。
- plotly 用于 3-D 绘图。
Titanic 数据集是一个 csv 文件,我们可以使用 read.csv 函数加载它。该数据集包含有关泰坦尼克号乘客的信息,包括以下列:
- 幸存者 — 指示乘客是否在泰坦尼克号坠机事故中幸存的标志。
- pclass——乘客的客票等级。
- 性别——乘客的性别。
- 年龄 – 年龄。
- sibsp——泰坦尼克号上的兄弟姐妹/配偶的数量。
- parch——泰坦尼克号上的父母/孩子人数。
- 票 – 票号。
- fare— Passenger fare.
- 客舱 — 乘客的客舱编号。
- 已登船 — 乘客的登船港。
我已经使用以下方法加载了它:
titanic <- read.csv(‘train.csv’)为简化起见,我将只使用来自 titanicdataframe 的原始列的子集——让我使用 dplyr 选择它们:
titanic <- titanic %>%
select(Fare, Age, Sex, Pclass, Survived, SibSp, Parch)让我们也将我们的数据拆分为训练一个测试——将 20% 的数据作为保留组:
# Splitting data into Train and Test
titanic['row_id'] = rownames(titanic)set.seed(123)
train_data <- titanic %>%
sample_frac(0.8)test_data <- titanic %>%
anti_join(train_data, by='row_id')# Drop row_id from both dataframes
train_data[,'row_id'] <- NULL
test_data[,'row_id'] <- NULL
尽管我们将在我们的超参数调整中使用交叉验证(稍后我会提到)——测试集将用于确保我们不会过度拟合我们的训练集或交叉验证集。对于决策树来说,这非常重要,因为它们很容易出现高方差。
在我们继续之前,让我们检查一下我们的数据预览:
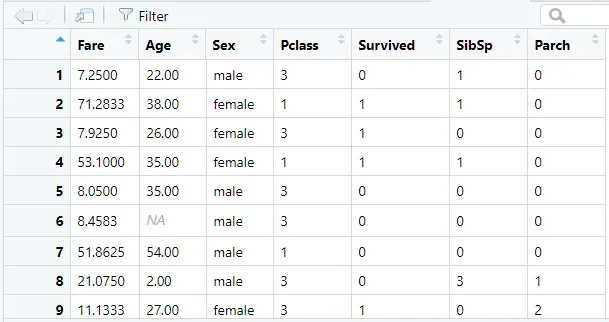
酷,我们的数据就位,让我们拟合我们的第一个决策树!
拟合第一决策树
对于决策树的第一个普通版本,我们将使用带有默认超参数的 rpart 包。
d.tree = rpart(Survived ~ .,
data=train_data,
method = 'class')由于我们没有指定超参数,我们使用 rpart 的默认值:[0]
- 我们的树可以下降到 30 层——maxdepth = 30;
- 节点中执行拆分的最小示例数为 20 — minsplit = 20 ;
- 一个终端节点的最小样本数为 7 — minbucket = 7;
- 拆分必须将树的“性能”(虽然不是那么直接,我们可以将“性能”视为 cp 的代理)至少增加 0.01 — cp = 0.01 ;
我们怎么知道这些是我们数据的最佳超参数?这些是随机选择,使用默认值是一个冒险的赌注。
也许我们可以进一步分离节点。或者,也许我们使用低样本基于低 minsplit 和 minbucket 做出决策。在继续之前,这是我们的树:
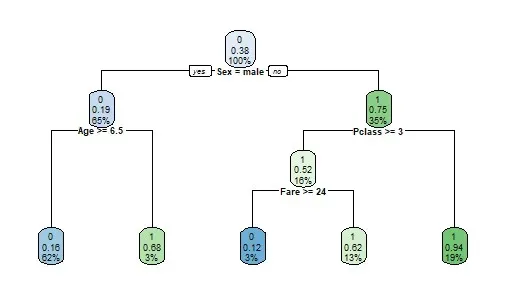
我们看到这是一棵比较浅的树,有 4 层。让我们检查测试集的准确性:
# Predict Values
predicted_values <- predict(d.tree, test_data, type = 'class')# Getting Accuracy
accuracy(test_data$Survived, predicted_values)
我们的准确率约为 79.21%。我们还能提高这个值吗?也许!调整超参数是我们可以探索的首批想法之一!
让我们首先手动设置它们——我们可以在 rpart 函数中使用 rpart.control 来覆盖默认超参数:
d.tree.custom = rpart(Survived~ .,
data=train_data,
method = 'class',
control = c(maxdepth = 5, cp=0.001))在这棵树上,我将 maxdepth 设置为 5,迫使我的树比我们上面看到的更深。此外,我还调整了 cp——让我们看看结果:
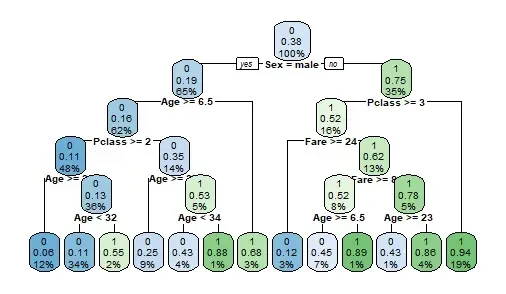
新树更深一些,包含更多规则——就性能而言,它的准确率约为 79.78%,比我们的香草版本好一点!
随着我们的准确性提高了几个点,我们的指标正在发生变化。从我们可以调整的整个超参数领域来看,其中一定有一些能够在测试集上产生最佳性能——对吧?我们是否必须手动尝试这些参数?
幸运的是,没有!尽管 rpart 不能让我们自动进行搜索,但我们有一个名为 mlr 的库可以帮助我们!
使用 MLR 进行超参数调整——调整一个参数
一件很酷的事情是,我们将在这里学到的东西对其他模型来说是广泛的。 mlrlibrary 使用与我们将学习调整随机森林、xgboost、SVM 等参数的方法完全相同的方法。
过去,您可能听说过 caret,一个著名的 R 数据科学库。尽管 caret 也有一些内置的超参数搜索,但 mlr 使我们能够更好地查看这些超参数的影响,减少“黑盒”——这就是我在这篇文章中使用 mlrin 的主要原因。
因此,mlr,R 库中的机器学习是 R 中一个很酷的人工智能包,它为我们提供了训练几个不同模型和执行调整的工具。正如我们所讨论的,优点之一是它可以让我们查看每个超参数对模型性能的影响。[0]
mlris getParamSet 中有一个方便的函数,它返回可用于特定模型的所有可调整参数——对于分类 rpart,我们可以调用 getParamSet(“classif.rpart”),它产生:
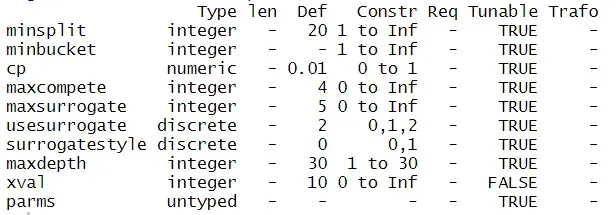
所有这些参数都可以使用 mlr 进行调整。让我们关注其中的 3 个—— minsplit 、 maxdepth 和 cp —— 仅从 maxdepth 开始。
在 constr 列上,我们可以看到可以调整的值范围——对于 maxdepth,我们可以从 1 到 30 的深度。
有一种简单的方法来拟合这 30 个不同版本的决策树并评估这些模型的准确性,这不是很有趣吗?这就是mlr所做的!
mlr 需要比普通的 rpartor 甚至插入符号更多的代码。首先,我们需要定义一个任务——在这种情况下,我定义了一个带有 train_data 和 target = ‘Survived 的分类任务:
d.tree.params <- makeClassifTask(
data=train_data,
target=”fraud”
)然后,我们需要创建参数网格以进行迭代——让我们从前面讨论过的单个参数慢慢开始。我们需要makeParamSet 并使用 makeDiscreteParam :
param_grid <- makeParamSet(
makeDiscreteParam(“maxdepth”, values=1:30))我在上面的代码中说明的是,我的树将迭代 30 个不同的 maxdepth 值,一个向量 (1:30),其中包含 1、2、3……、30 作为输入超参数的值。
然后,我们需要做三件事——初始化控制网格实验,选择交叉验证方法并选择用于评估结果的度量:
# Define Grid
control_grid = makeTuneControlGrid()# Define Cross Validation
resample = makeResampleDesc("CV", iters = 3L)# Define Measure
measure = acc
交叉验证是一种改进决策树结果的方法。我们将在示例中使用三重交叉验证。对于度量,我们将使用准确度 (acc)。[0]
可以了,好了 !是时候将所有内容都输入到 magictuneParams 函数中了,这将启动我们的超参数调整!
set.seed(123)
dt_tuneparam <- tuneParams(learner=’classif.rpart’,
task=d.tree.params,
resampling = resample,
measures = measure,
par.set=param_grid,
control=control_grid,
show.info = TRUE)当您运行上面的代码时,我们的超参数搜索将开始执行! show.info = TRUE 将输出执行的反馈:
[Tune-x] 1: maxdepth=1
[Tune-y] 1: acc.test.mean=0.7895909; time: 0.0 min
[Tune-x] 2: maxdepth=2
[Tune-y] 2: acc.test.mean=0.7881845; time: 0.0 min
[Tune-x] 3: maxdepth=3
[Tune-y] 3: acc.test.mean=0.8008132; time: 0.0 min
...每个 maxdepth 生成一个 acc.test.mean,即交叉验证中使用的几个数据集的平均值。 mlr 还让我们使用 generateHyperParsEffectData 评估结果:
result_hyperparam <- generateHyperParsEffectData(dt_tuneparam, partial.dep = TRUE)我们可以使用 ggplot 绘制精度的演变:
ggplot(
data = result_hyperparam$data,
aes(x = maxdepth, y=acc.test.mean)
) + geom_line(color = 'darkblue')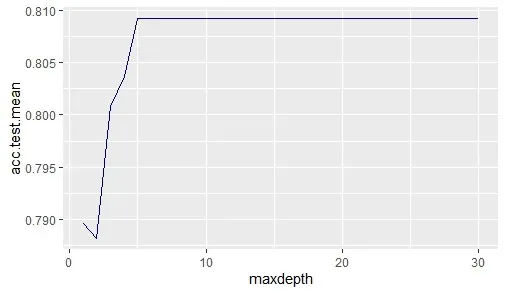
查看我们的图,我们了解到在深度为 5 之后,对准确性的影响是微不足道的,差异非常小。让我们确认 tuneParams 函数选择的最佳模型是什么——我们可以通过直接调用 dt_tuneparam 对象来检查:
Tune result:
Op. pars: maxdepth=11
f1.test.mean=0.9985403调整结果选择 19 maxdepth 作为最佳参数,只是因为差异很小——不过,让我们使用对象 dt_tuneparam$x 来拟合我们的最佳参数,以获取保存的超参数并使用 setHyperPars 存储它们:
best_parameters = setHyperPars(
makeLearner(“classif.rpart”),
par.vals = dt_tuneparam$x
)best_model = train(best_parameters, dt_task)
train 将使用 best_parameters 对象中保存的超参数拟合决策树。
运行上面的代码后,我们得到了一个拟合树,其中包含从 best_model 上的网格搜索返回的最佳超参数。为了在我们的测试集上评估这个模型,我们需要创建一个新的 makeClassifTask 指向测试数据:
d.tree.mlr.test <- makeClassifTask(
data=test_data,
target=”Survived”
)预测和检查 test_data 的准确性:
results <- predict(best_model, task = d.tree.mlr.test)$data
accuracy(results$truth, results$response)我们的准确率约为 79.21%,与我们的香草版本相同。所以……可能,我们对 cp 参数的调整是提高模型性能的诀窍。
问题是..在这个例子中,我们保持其他参数不变,这是否意味着我们只能一一调整我们的超参数?没有!
使用 mlr ,我们可以同时调整整个参数环境,只需对我们的代码进行一点微调!让我们这样做。
Tweaking Multiple Parameters
调整多个超参数很容易!还记得我们为网格搜索创建的 param_grid 对象吗?让我们回忆一下:
param_grid <- makeParamSet(
makeDiscreteParam(“maxdepth”, values=1:30))如果我在 makeParamSet 函数中添加新参数,我将添加将在搜索中组合的新参数 – 例如,让我们将 cp 和 minsplit 添加到我们的景观中:
param_grid_multi <- makeParamSet(
makeDiscreteParam(“maxdepth”, values=1:30),
makeNumericParam(“cp”, lower = 0.001, upper = 0.01),
makeDiscreteParam(“minsplit”, values=1:30)
)makeNumericParam 创建数字参数(例如包含小数位的 cp)——我们可以在 getParamSet 函数中检查哪些超参数是离散的或数字的(请记住,可以使用 makeDiscreteParam 调用整数)。
我们如何训练这种多参数搜索?通过将我们的 param_grid_multi 输入到 tuneParams 函数!
dt_tuneparam_multi <- tuneParams(learner=’classif.rpart’,
task=d.tree.mlr,
resampling = resample,
measures = measure,
par.set=param_grid_multi,
control=control_grid,
show.info = TRUE)当我们训练更多的超参数时,会产生计算成本。您会注意到 dt_tuneparam_multi 将比 dt_tuneparam 搜索花费更多时间,因为我们将在数据中拟合近 3000(!) 棵树。
在搜索结束时,您可能会得到类似于以下输出的内容:
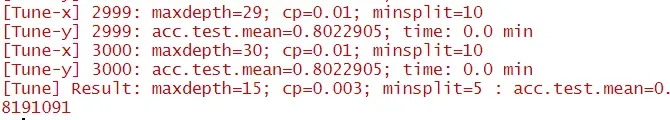
在 [Tune] 输出中,我们有搜索的最佳参数:
- a maxdepth of 15.
- a cp of 0.003.
- a minsplit of 5.
这种超参数的组合在交叉验证中产生了大约 82% 的准确率,还不错!
让我们提取最佳参数,用它们训练一棵新树,然后在我们的测试集上查看结果:
# Extracting best Parameters from Multi Search
best_parameters_multi = setHyperPars(
makeLearner(“classif.rpart”, predict.type = “prob”),
par.vals = dt_tuneparam_multi$x
)best_model_multi = train(best_parameters_multi, d.tree.mlr)# Predicting the best Model
results <- predict(best_model_multi, task = d.tree.mlr.test)$dataaccuracy(results$truth, results$response)
我们在测试集中的准确率为 81.46%!仅通过调整这些参数,我们就能够将基线准确度提高 2 个百分点,这是一个极好的结果!
作为最后一点并帮助您可视化我们所做的工作,让我们绘制网格搜索结果样本的准确性结果:
# Extracting results from multigrid
result_hyperparam.multi <- generateHyperParsEffectData(dt_tuneparam_multi, partial.dep = TRUE)# Sampling just for visualization
result_sample <- result_hyperparam.multi$data %>%
sample_n(300)hyperparam.plot <- plot_ly(result_sample,
x = ~cp,
y = ~maxdepth,
z = ~minsplit,
marker = list(color = ~acc.test.mean, colorscale = list(c(0, 1), c(“darkred”, “darkgreen”)), showscale = TRUE))
hyperparam.plot <- hyperparam.plot %>% add_markers()
hyperparam.plot
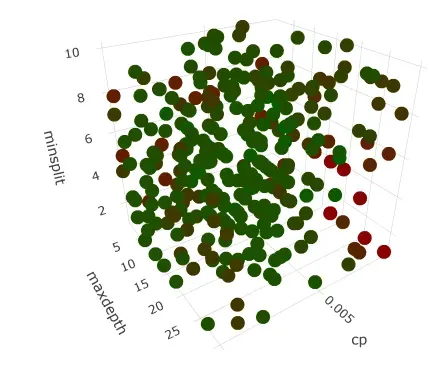
在 y 轴上,我们有 minsplit 。在 x 轴上我们有 maxdepth 在 z 轴上我们有 cp 。
每个点都是一个实验(超参数的组合),颜色与该实验的准确度结果相关。红点表示精度较低。绿点表示更好的性能。
在 3-d 图中有一个明显的红色区域,我们看到 cp 的结果不是很好——让我旋转它以获得更好的视图:
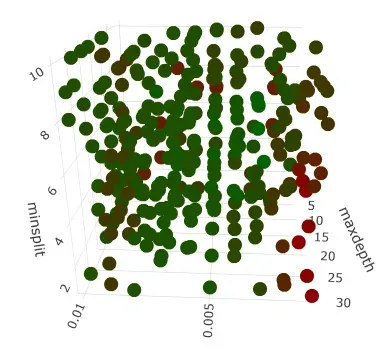
请注意,非常低的 cp 会产生更差的性能,尤其是与低 minsplit 结合使用时!
可视化我们的超参数搜索结果让我们可以很好地鸟瞰我们的训练过程是如何表现的。
如果您想查看上面情节的交互式版本,请点击此链接![0]
感谢您花时间阅读这篇文章!我希望你已经欣赏它,你现在可以理解如何使用 R 训练超参数。
超参数可以建立或破坏模型,作为数据科学家,我们需要知道如何用几行代码有效地调整它们。如果您使用 R,mlr 可能是执行此常见机器学习任务的绝佳选择!
我在 Udemy 上设置了 R 简介和学习数据科学的训练营。这两门课程都是为初学者量身定制的,我很想有你在身边![0][1]

这是这篇文章中代码的一个小要点:
titanic <- read.csv('train.csv')
library(dplyr)
library(rpart)
library(rpart.plot)
library(Metrics)
library(mlr)
library(ggplot2)
library(plotly)
# Sample Fraud Data to speed up execution
set.seed(123)
# Subset Columns for Decision Tree
titanic <- titanic %>%
select(Fare, Age, Sex, Pclass, Survived, SibSp, Parch)
# Splitting data into Train and Test
titanic['row_id'] = rownames(titanic)
set.seed(123)
train_data <- titanic %>%
sample_frac(0.8)
test_data <- titanic %>%
anti_join(train_data, by='row_id')
# Drop row_id from both dataframes
train_data[,'row_id'] <- NULL
test_data[,'row_id'] <- NULL
# Building our first decision tree
d.tree = rpart(Survived ~ .,
data=train_data,
method = 'class')
# Plotting our Tree
rpart.plot(d.tree, cex=0.55)
# Predict Values
predicted_values <- predict(d.tree, test_data, type = 'class')
# Getting Accuracy
accuracy(test_data$Survived, predicted_values)
# Building our d.tree with custom paremeters
d.tree.custom = rpart(Survived~ .,
data=train_data,
method = 'class',
control = c(maxdepth = 5, cp=0.001))
rpart.plot(d.tree.custom, cex=0.6)
# Predict test set data
predicted_values.custom <- predict(d.tree.custom, test_data, type = 'class')
# Accuracy of Custom D.Tree
accuracy(test_data$Survived, predicted_values.custom)
# Hyperparameter Tuning training with mlr
getParamSet("classif.rpart")
d.tree.mlr <- makeClassifTask(
data=train_data,
target="Survived"
)
# Search Parameter for Max Depth
param_grid <- makeParamSet(
makeDiscreteParam("maxdepth", values=1:30))
# Define Grid
control_grid = makeTuneControlGrid()
# Define Cross Validation
resample = makeResampleDesc("CV", iters = 3L)
# Define Measure
measure = acc
set.seed(123)
dt_tuneparam <- tuneParams(learner='classif.rpart',
task=d.tree.mlr,
resampling = resample,
measures = measure,
par.set=param_grid,
control=control_grid,
show.info = TRUE)
# Extracting results
result_hyperparam <- generateHyperParsEffectData(dt_tuneparam, partial.dep = TRUE)
# Plotting Accuracy Score across different maxdepth
ggplot(
data = result_hyperparam$data,
aes(x = maxdepth, y=acc.test.mean)
) + geom_line(color = 'darkblue')
dt_tuneparam
# Pick Up Best Params and train them
best_parameters = setHyperPars(
makeLearner("classif.rpart", predict.type = "prob"),
par.vals = dt_tuneparam$x
)
best_model = train(best_parameters, d.tree.mlr)
d.tree.mlr.test <- makeClassifTask(
data=test_data,
target="Survived"
)
# Predicting the best Model
results <- predict(best_model, task = d.tree.mlr.test)$data
accuracy(results$truth, results$response)
# Tweaking multiple hyperparameters
param_grid_multi <- makeParamSet(
makeDiscreteParam("maxdepth", values=1:30),
makeNumericParam("cp", lower = 0.001, upper = 0.01),
makeDiscreteParam("minsplit", values=1:10)
)
dt_tuneparam_multi <- tuneParams(learner='classif.rpart',
task=d.tree.mlr,
resampling = resample,
measures = measure,
par.set=param_grid_multi,
control=control_grid,
show.info = TRUE)
# Extracting best Parameters from Multi Search
best_parameters_multi = setHyperPars(
makeLearner("classif.rpart", predict.type = "prob"),
par.vals = dt_tuneparam_multi$x
)
best_model_multi = train(best_parameters_multi, d.tree.mlr)
# Predicting the best Model
results <- predict(best_model_multi, task = d.tree.mlr.test)$data
accuracy(results$truth, results$response)
# Extracting results from multigrid
result_hyperparam.multi <- generateHyperParsEffectData(dt_tuneparam_multi, partial.dep = TRUE)
# Sampling just for visualization
result_sample <- result_hyperparam.multi$data %>%
sample_n(300)
hyperparam.plot <- plot_ly(result_sample,
x = ~cp,
y = ~maxdepth,
z = ~minsplit,
marker = list(color = ~acc.test.mean, colorscale = list(c(0, 1), c("darkred", "darkgreen")), showscale = TRUE))
hyperparam.plot <- hyperparam.plot %>% add_markers()
hyperparam.plot
数据集许可证:本文中使用的数据集可在 https://www.openml.org/d/40945 公开使用[0]
文章出处登录后可见!
