参考:https://www.bilibili.com/video/BV1HL4y1M7ue?spm_id_from=333.337.search-card.all.click
一、前期探索
前言:用别人已经分类好的结果来分类,小编也不知道自己在干什么呢!~_~
前期准备:把2017-2019年的分类结果按掩膜提取到自己的研究区,并用重分类提取出玉米、大豆栅格。再取交集找到这三年都种玉米或都种大豆的地块。
先上一下这部分结果:
17年玉米种植区分布:

18年玉米种植区分布:
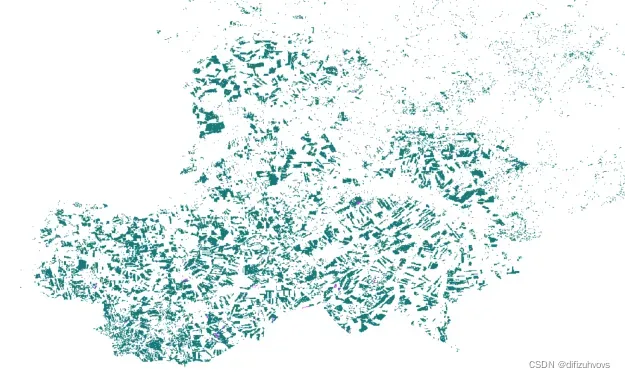
19年玉米种植区分布:

取交集后:
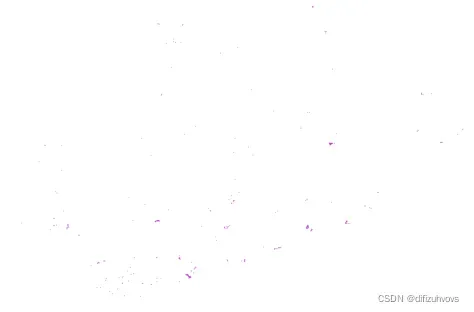
呃呃。。。怎么重叠部分这么少呢~_~相信大家懂得都懂,这里就看破不说破……………………………………………………
二、上传边界及样本参考
前面的步骤忽略不计。只选用2019年的分类结果,上传到GEE,10m分辨率的tif传了一下午也失败了,于是重采样到100m上传。
结果原来的4类在GEE中展示只有一类。如下图。
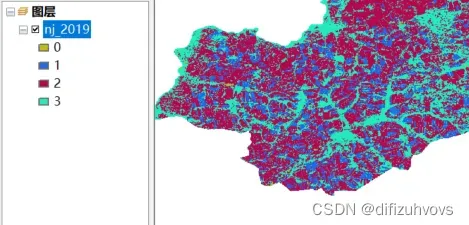
arcmap中的分类结果:
放到GEE中展示:
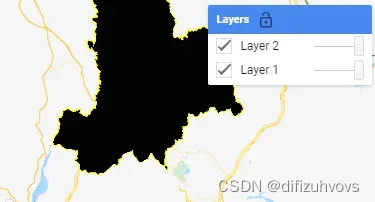
所以还是得分成“玉米”、“大豆”两类上传。
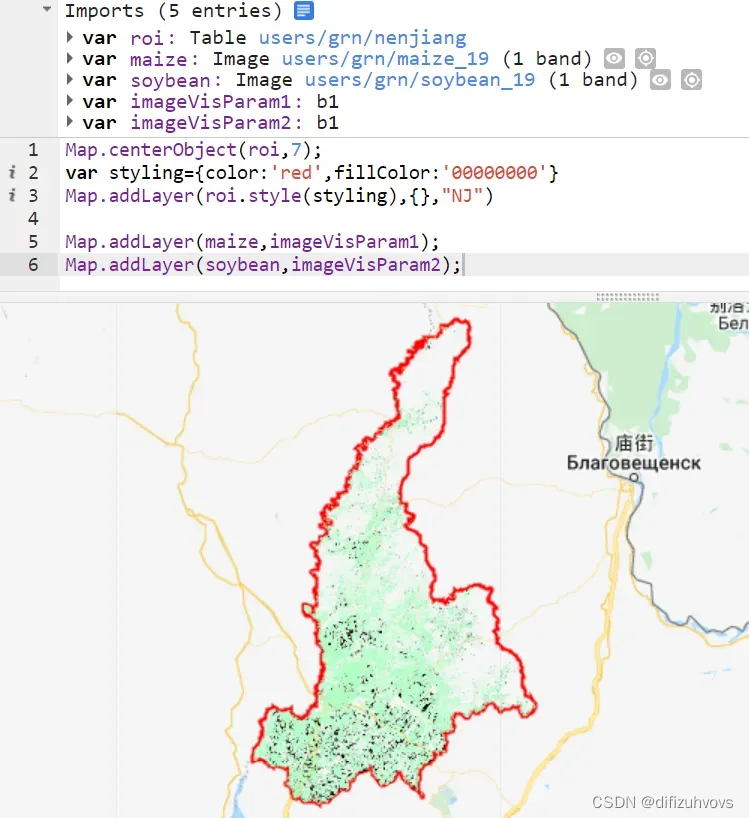
展示我们的研究区范围及分类结果。
Map.centerObject(roi,7);
var styling={color:'red',fillColor:'00000000'}
Map.addLayer(roi.style(styling),{},"NJ")
Map.addLayer(maize,imageVisParam1);
Map.addLayer(soybean,imageVisParam2);
三、建立样本数据集
在分类结果上选取玉米种植区样本60个,大豆种植区样本60个,“其它”60个。
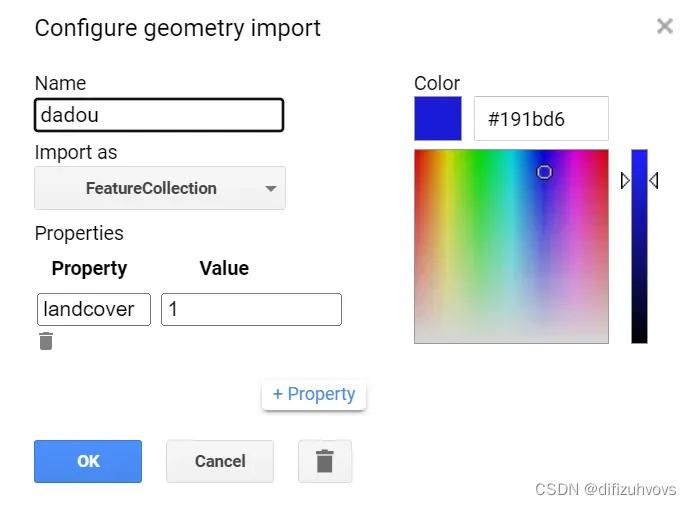
样本数据集建立方法是点击这个“添加标记”,然后点击这个设置按钮修改属性,如下三图所示。
标好之后导出,免得丢失。

Export.table.toDrive({
collection:dadou,
description: "dadou",
fileNamePrefix: "dadou",
folder:'sampleShp',
fileFormat: "SHP"
});
Export.table.toDrive({
collection:yumi,
description: "yumi",
fileNamePrefix: "yumi",
folder:'sampleShp',
fileFormat: "SHP"
});
Export.table.toDrive({
collection:qita,
description: "qita",
fileNamePrefix: "qita",
folder:'sampleShp',
fileFormat: "SHP"
});
四、利用随机森林分类
Map.centerObject(roi,7);
// var styling={color:'red',fillColor:'00000000'}
// Map.addLayer(roi.style(styling),{},"NJ")
// Map.addLayer(maize,imageVisParam1);
// Map.addLayer(soybean,imageVisParam2);
//选择需要裁剪的矢量数据
var aoi = roi;
//加载矢量边框,以便于在边界内选取样本点
var empty = ee.Image().toByte();
var outline = empty.paint({
featureCollection:aoi, // 行政边界命名为fc
color:0, //颜色透明
width:3 //边界宽度
});
Map.addLayer(outline, {palette: "ff0000"}, "outline");
//Function to mask the clouds in Sentinel-2
function maskS2clouds(image) {
var qa = image.select('QA60');
// Bits 10 and 11 are clouds and cirrus, respectively.
var cloudBitMask = 1 << 10;
var cirrusBitMask = 1 << 11;
// Both flags should be set to zero, indicating clear conditions.
var mask = qa.bitwiseAnd(cloudBitMask).eq(0)
.and(qa.bitwiseAnd(cirrusBitMask).eq(0));
return image.updateMask(mask).divide(10000);
}
//Build the Sentinel 2 collection, filtered by date, bounds and percentage of cloud cover
var dataset = ee.ImageCollection('COPERNICUS/S2_SR')
.filterDate('2019-01-01','2019-12-31')
.filterBounds(aoi)
.filter(ee.Filter.lt('CLOUDY_PIXEL_PERCENTAGE',10))
.map(maskS2clouds);
print("Sentinel 2 Image Collection",dataset);
var dem = ee.Image("NASA/NASADEM_HGT/001")
// Construct Classfication Dataset
// RS Index Cacluate(NDVI\NDWI\EVI\BSI)
var add_RS_index = function(img){
var ndvi = img.normalizedDifference(['B8', 'B4']).rename('NDVI').copyProperties(img,['system:time_start']);
var ndwi = img.normalizedDifference(['B3', 'B8']).rename('NDWI').copyProperties(img,['system:time_start']);
var evi = img.expression('2.5 * ((NIR - RED) / (NIR + 6 * RED - 7.5 * BLUE + 1))',
{
'NIR': img.select('B8'),
'RED': img.select('B4'),
'BLUE': img.select('B2')
}).rename('EVI').copyProperties(img,['system:time_start']);
var bsi = img.expression('((RED + SWIR1) - (NIR + BLUE)) / ((RED + SWIR1) + (NIR + BLUE)) ',
{
'RED': img.select('B4'),
'BLUE': img.select('B2'),
'NIR': img.select('B8'),
'SWIR1': img.select('B11')
}).rename('BSI').copyProperties(img,['system:time_start']);
var ibi = img.expression('(2 * SWIR1 / (SWIR1 + NIR) - (NIR / (NIR + RED) + GREEN / (GREEN + SWIR1))) / (2 * SWIR1 / (SWIR1 + NIR) + (NIR / (NIR + RED) + GREEN / (GREEN + SWIR1)))', {
'SWIR1': img.select('B11'),
'NIR': img.select('B8'),
'RED': img.select('B4'),
'GREEN': img.select('B3')
}).rename('IBI').copyProperties(img,['system:time_start']);
return img.addBands([ndvi, ndwi, evi, bsi, ibi]);
};
var dataset = dataset.map(add_RS_index);
var bands = ['B2','B3','B4','B5','B6','B7','B8','B8A','B11','NDVI','NDWI','BSI'];
var imgcol_median = dataset.select(bands).median();
var aoi_dem = dem.select('elevation').clip(aoi).rename('DEM');
var construct_img = imgcol_median.addBands(aoi_dem).clip(aoi);
//分类样本
var train_points = dadou.merge(yumi).merge(qita);
//精度评价
var withRandom = train_points.randomColumn('random');//样本点随机的排列
var split = 0.7;
var trainingPartition = withRandom.filter(ee.Filter.lt('random', split));//筛选70%的样本作为训练样本
var testingPartition = withRandom.filter(ee.Filter.gte('random', split));//筛选30%的样本作为测试样本
var training= construct_img.sampleRegions({
collection: trainingPartition,
properties: ['landcover'],
scale: 10
});
var validation= construct_img.sampleRegions({
collection: testingPartition,
properties: ['landcover'],
scale: 10
});
//分类方法选择随机森林
var rf = ee.Classifier.smileRandomForest({
numberOfTrees: 50,
bagFraction: 0.8
}).train({
features: training,
classProperty: 'landcover',
// inputProperties: inputbands
});
//对哨兵数据进行随机森林分类
var img_classfication = construct_img.classify(rf);
//运用测试样本分类,确定要进行函数运算的数据集以及函数
var test = validation.classify(rf);
//计算混淆矩阵
var confusionMatrix = test.errorMatrix('landcover', 'classification');
print('confusionMatrix',confusionMatrix);//面板上显示混淆矩阵
print('overall accuracy', confusionMatrix.accuracy());//面板上显示总体精度
print('kappa accuracy', confusionMatrix.kappa());//面板上显示kappa值
Map.addLayer(aoi);
Map.addLayer(img_classfication.clip(aoi), {min: 1, max: 3, palette: ['orange', 'blue', 'green']});
var class1=img_classfication.clip(aoi)
// //导出分类图
// Export.image.toDrive({
// image: class1,
// description: 'rfclass',
// fileNamePrefix: 'rf', //文件命名
// folder: "class", //保存的文件夹
// scale: 10, //分辨率
// region: aoi, //研究区
// maxPixels: 1e13, //最大像元素,默认就好
// crs: "EPSG:4326" //设置投影
// });
一开始用的参考视频里小姐姐的代码,但是总体分类精度和kappa系数一直显示为1,我还以为是我样本选得不够多,于是样本各增加到100,结果还是都为1,原来是代码里有些小错,改完后结果如下图。
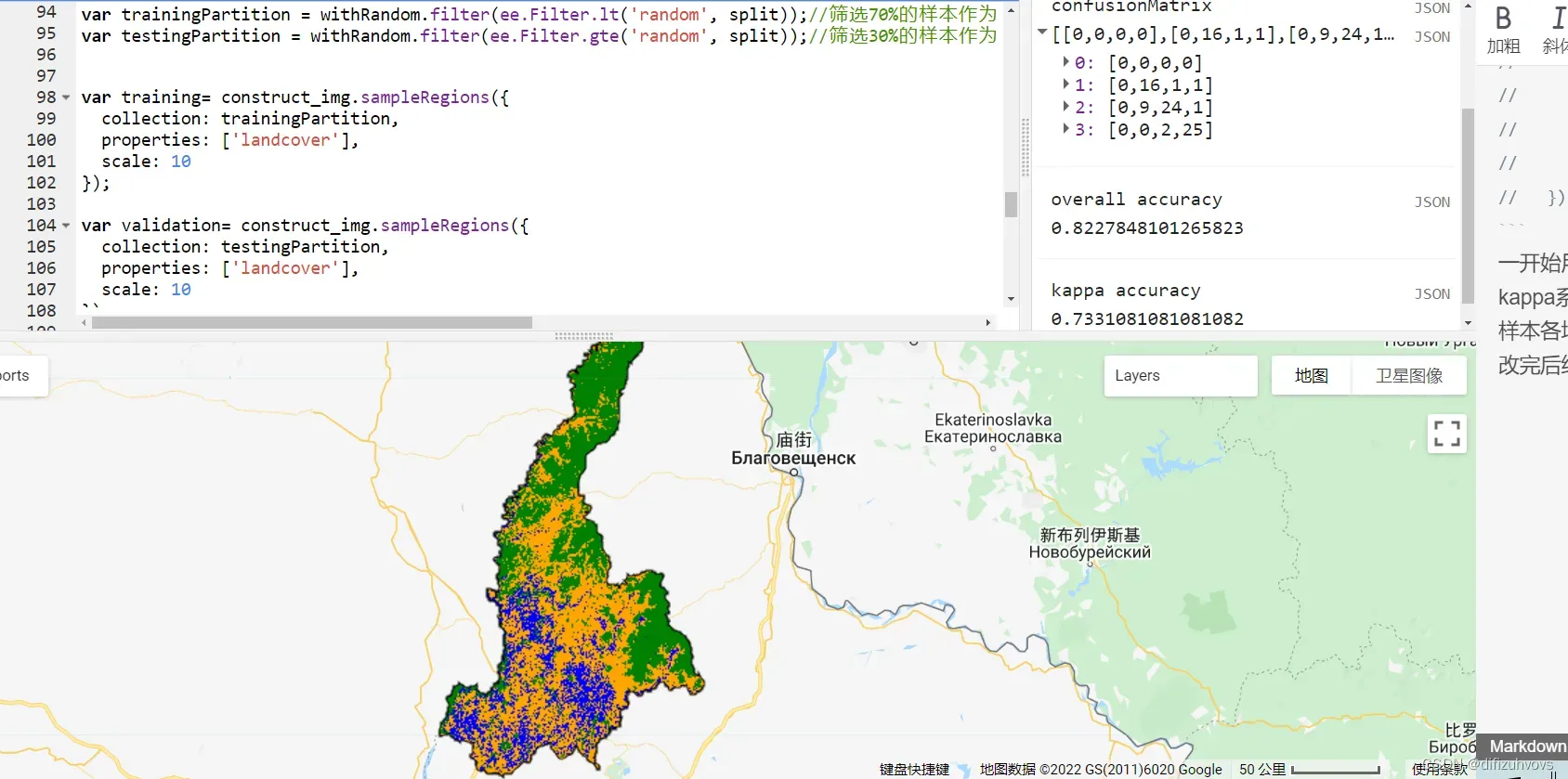
就很玄学,呵呵~
五、220511换一个试点地区
今天做就快好多了,在arcmap中进行的处理总共就这几步:

分类结果:
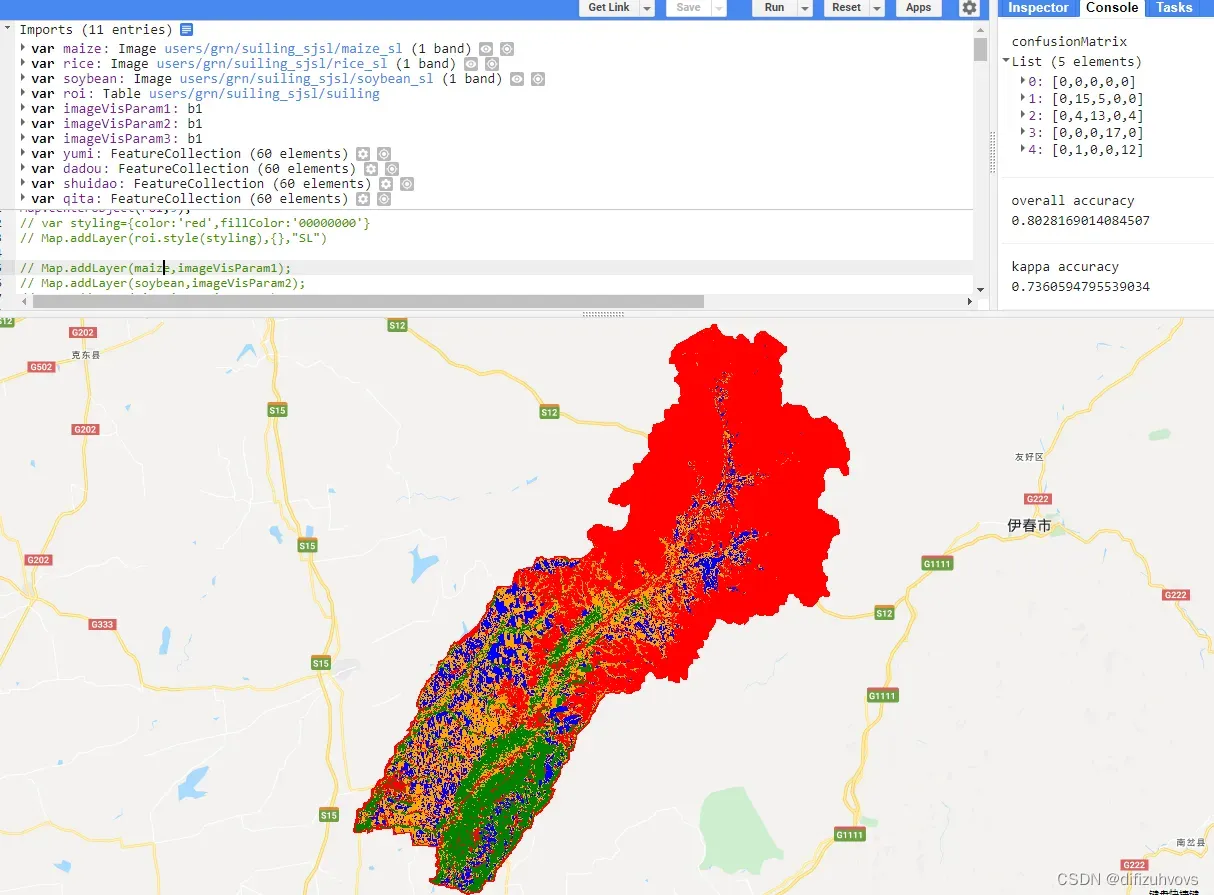
一个小时就能有个交代了=。=
文章出处登录后可见!
已经登录?立即刷新
